|
|
|
发布时间: 2022-06-16 |
数据挖掘和信息交互 |
|
|
|
收稿日期: 2022-01-10; 修回日期: 2022-03-16; 预印本日期: 2022-03-23
基金项目: 国家自然科学基金项目(82001450);博士后创新人才支持计划(BX20200364);科技创新2030-“脑科学与类脑研究”重大项目(2021ZD0200201)
作者简介:
罗娜,1990年生,女,副研究员,主要研究方向为跨模态数据融合方法及其应用。E-mail: luona2015@ia.ac.cn
宋明,男,研究员,主要研究方向为多模态脑成像及其在脑科学和脑疾病中的应用。E-mail: msong@nlpr.ia.ac.cn 杨正宜,男,副研究员,主要研究方向为医学影像分析和机器人应用。E-mail: zhengyi.yang@nlpr.ia.ac.cn 蒋田仔,通信作者,男,研究员,主要研究方向为跨模态多尺度脑网络组图谱、基于脑网络组图谱的脑机融合和脑疾病早期预测与精准治疗。E-mail: jiangtz@nlpr.ia.ac.cn *通信作者: 蒋田仔 jiangtz@nlpr.ia.ac.cn
中图法分类号: TP399
文献标识码: A
文章编号: 1006-8961(2022)06-2036-21
|
摘要
脑图谱是研究脑结构和功能及脑疾病的基础,不同类型的脑图谱从不同角度提供了脑的组织模式或连接信息。随着图像采集和生物检测技术的发展,不同模态的脑影像和生物组学数据迅速增长。相较于单模态,多模态融合数据能够同时考察不同模态数据间的多元化信息,挖掘蕴含的未知新信息。因此,开展跨模态脑图谱数据融合研究有助于更全面地理解大脑的结构和功能,并辅助加深对脑发育、老化和病变机理的理解。本文根据参与融合的模态是否具有空间信息,将近年来有代表性的跨模态脑图谱融合技术分为脑影像融合和脑数据融合两大类。脑影像融合是指对宏观脑影像(磁共振等)和组织学脑影像(胞体染色、轴突染色等)等具有空间信息的数据进行融合,构建涵盖脑结构和功能信息的跨模态多尺度脑图谱, 为研究宏观特征的介观机制以及介观特征的宏观表征提供了重要途径。脑数据融合是指对缺乏脑空间信息的生物大数据,包括基因组、电生理、认知和行为等,利用脑图谱提供精细空间信息,挖掘高维、异构生物大数据蕴含的信息,明确脑图谱的生理意义,并提升其应用价值。本文将针对这两类融合类型阐述国内外有代表性的研究进展,并对比国内外研究现状的差异。此外,为促进跨模态脑图谱数据融合领域的交流和发展,总结了部分有代表性的大样本公开数据集。最后讨论了当前该领域待解决的问题以及未来的发展趋势。
关键词
脑图谱; 跨模态; 空间信息; 脑影像; 多组学; 公开数据集
Abstract
The human brain is the most complex network system in the world. Different types of brain atlas provide brain tissue or connection information from different aspects. With the development of image acquisition and biological detection technology, researchers reveal that combing multi-modal brain data is able to provide more information through exploiting the rich multimodal information that exits, for example it can inform us about how brain structure shapes brain function, in which way they are impacted by psychopathology and which functional or structural aspects of physiology could drive human behavior and cognition. Through summarizing the representative multi-modal brain fusion technologies in recent years, we first divide them into two categories in this review, brain-imaging fusion and brain-data fusion. 1)Brain-imaging fusion refers to the fusion of data with fine spatial information such as brain images (magnetic resonance imaging, etc.) and histological images (cell body staining, axon staining, etc.), through which could help construct a multi-modal multi-scale brain map covering both structure and function information. For example, the Julich-Brain Atlas, a three-dimensional atlas containing cytoarchitectonic maps of cortical areas and subcortical nuclei, is built through the fusion of magnetic resonance imaging and histological slices, allowing comparison of functional activations, networks, genetic expression patterns, anatomical structures, and other data obtained across different studies in a common stereotaxic reference space. The multiscale brain atlas brings together data from these different levels of nervous system organization to form a better understanding of between-scale relationships of brain structure, function, and behavior in health and disease. 2)Brain-data fusion refers to applying brain atlas with spatial information to fuse biological big data without spatial information (genome, electrophysiology, cognition and behavior, etc.), through which could help make up the shortcomings from single modality and capitalize on joint information among modalities to dig out new information. For example, combing brain atlas with electroencephalogram data can simultaneously extract signals with high spatial and temporal resolution. For each fusion type discussed above, we elaborate the representative research progress at home and abroad in recent years, as well as their comparison in the current review. Then, in order to promote the development of this filed, we introduce several multi-modal datasets in the community, i.e., UK Biobank, Human Connectome Project(HCP), Chinese Imaging Genetics(CHIMGEN), Adolescent Brain Cognitive Development(ABCD), Philadelphia Neurodevelopmental Cohort(PNC), IMAGEN, Pediatric Imaging, Neurocognition, and Genetics(PING), Autism Brain Imaging Data Exchang(ABIDE), Alzheimer's Disease Neuroimaging Initiative(ADNI), ADHD-200, PRIMatE Data Exchange (PRIME-DE), National Chimpanzee Brain Resource(NCBR), the Allen Atlas and EBRAINS Reference Atlas. Finally, we discuss the important problems to be solved and the future direction in this field. The common challenge of this filed is how to integrate the information from different scales and modalities to build a multi-modal and cross-scale brain atlas, and how to analyze the neural mechanism of major brain diseases based on the new brain atlas, and forming a new paradigm for brain disorder diagnosis and treatment paradigm at last. To achieve this major goal, 1)the acquisition of high-quality big data is the important foundation, which rely on the development of acquisition equipments. 2)The high resolution of data (i.e., cell and neuron level)raises large storaging and quick processing demands. 3)Developing new fusion methods based on artificial intelligence is also one of the urgent issues in multi-modal fusion research.
Key words
brain atlas; cross-modal; spatial information; brain imaging; multi-omics; public dataset
0 引言
脑是人体最为复杂的器官,研究脑不仅能够帮助人们理解认知、思维、意识和语言等脑功能的基本原理,解析其神经机理,还对人类的健康至关重要,并辅助减轻脑疾病带来的家庭和社会负担。然而,要揭示其复杂的工作原理,最首要的是全面解析其网络结构,即构建能为复杂脑功能研究提供“导航”的脑图谱。因此,脑图谱是研究脑结构和功能的重要工具,对脑疾病防治、智能技术发展也具有引导作用(左年明和蒋田仔,2020)。此外,生物医学成像技术的发展使得获取多样的脑结构和功能信息成为可能,但是单一的成像技术通常只能获得脑组织片面的结构或功能信息,例如临床广泛采用的磁共振成像,单独的成像序列可以获得mm尺度的组织结构或血氧水平变化(但无法同时获得,需要在不同时间点分别成像),却不能直接刻画神经元电活动的信息;同样,临床脑电技术可以弥补上述缺点,但是它们本身不能获得解剖结构信息,并且空间分辨率较低。因此,融合多模态、跨尺度的生物医学图像构建全新的脑图谱并以脑图谱作为约束融合更多异构的生物大数据,是深入理解脑结构和功能的必经之路。
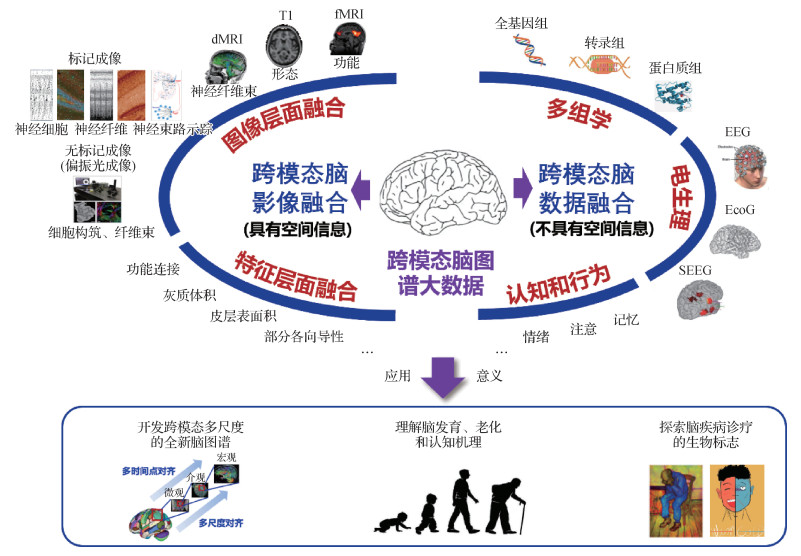
本文通过总结近年来有代表性的跨模态脑图谱融合研究,将其分为(如图 1所示):1)跨模态脑影像融合:借助高速发展的多模态成像技术,直接观察微观细胞结构的形态、连接等,再通过发展各种图像分割、配准和融合技术,来解析从介观神经元、细胞构筑到宏观脑结构联合和功能网络的分区边界,构建跨模态多尺度脑区成像信息的脑图谱关联关系,这类融合研究主要实现脑图谱技术在空间和时间维度的不断革新;2)跨模态脑数据融合:脑图谱为多组学、电生理、认知和行为等缺乏空间信息的多组学信息提供重要的定位基准,研究者们通过发展各种基于机器学习和回归模型的跨模态数据融合方法来探究认知和行为的脑影像基础及其底层生物学基础,以期通过这类融合研究辅助理解底层模态的生物过程是如何影响或改变宏观脑区的结构,最终导致大脑的失调、认知功能损伤和病变(van den Heuvel等,2019)。本文的重要意义如下:
1) 启发跨模态脑图谱数据融合的新方法。通过梳理、归纳和对比现有跨模态融合方法,帮助初学者快速了解和熟悉这个领域,同时通过总结未来该领域的发展方向,促进图像领域多学科交叉,开发更多跨模态脑图谱融合新方法。
2) 促进脑结构、功能和认知机理的探索。通过综述现有跨模态融合方法在脑区分割、多组学数据融合以及认知预测等方面的研究,有助于拓展跨模态融合方法在脑认知机理探索领域的应用,为解析脑的发展变化规律提供新工具。
3) 辅助临床发展脑疾病客观脑影像标记。通过总结不同跨模态融合方法及临床应用研究,有助于研究者通过结合实际临床问题,选择合适的跨模态融合方法,促进解决卡脖子的关键科学问题和技术瓶颈,为重大脑疾病的诊断和精准治疗提供科学依据。
1 国内外研究现状
1.1 跨模态脑影像融合
1.1.1 脑图谱图像层面融合
人脑图谱的绘制可以追溯到20世纪初,Brodmann(1909)利用尼氏染色人脑切片进行了基于细胞构筑的脑分区,由于该图谱分区粗糙且样本量少,已经不能满足当下的脑科学研究。中国科学院自动化研究所脑网络组研究中心2009年提出利用弥散磁共振成像获得的脑结构连接信息进行脑图谱绘制的思想,在2016年成功绘制出全新的人类脑图谱——脑网络组图谱,脑网络组图谱不仅包括精细的脑区划分,也包括脑区之间的连接图谱(Fan等,2016)。在此基础上,2020年该团队发现脑结构连接在多种功能领域上对脑功能激活的预测能力都超过随机对照模型,脑结构连接和功能关系在皮层上存在着层级结构,而且这种脑结构连接和功能激活关系的层级结构还与多种其他的脑组织结构有关,如髓鞘化、功能灵活性和功能个体差异等(Wu等,2020),这些发现系统地阐明了脑结构连接和功能激活之间的关系,不仅为脑网络组图谱绘制方法的合理性提供了重要的科学证据,也为理解脑的结构和功能组织提供了新见解。同样,为了捕捉不同模态的关联性,美国华盛顿大学医学院的David C Van Essen团队基于人类连接组项目(Human Connectome Project,HCP)提供的多模态数据集,采用半自动的分割技术,在210个健康年轻成人上分别进行皮层构筑、脑功能、脑连接和脑拓扑4种模态的边界划分,如果两个以上模态在同一个位置存在边缘分割,就将这一边缘定义为多模态脑图谱的脑区边界,由此泛化到全脑得到跨模态脑图谱(Glasser等,2016)。为了进一步完善皮下核团的分割精度,2020年9月,澳大利亚墨尔本大学的研究团队通过分析1 000多名健康成年人的高分辨率功能磁共振脑影像,成功绘制了一份迄今为止最为精细的人脑皮层下核团图谱(Tian等,2020)。这些皮层下核团主要负责调节和控制大脑皮层,以及与身体其他部分之间快速、海量的信息交换,虽然这些皮层下核团与大脑皮层在空间上相距甚远,但它们内部的信息交换以及它们与大脑皮层各区域之间紧密的连接和功能协同,促成了初级感觉运动功能、高级认知功能以及神经心理活动的高度协调统一。
虽然基于磁共振影像(magnetic resonance imaging,MRI)等构建的立体脑图谱具有良好的3维空间信息,可以有效地区分不同脑区的边界并计算不同脑区之间的结构连接等,但其分辨率为mm级,不能在神经元/细胞分辨水平真实反映脑结构与功能活动。近年来,随着超薄切片技术、染色技术以及显微观察技术的进步,基于细胞构筑的脑解剖结构研究得到了进一步的发展,特别是随着活体影像采集设备和计算机图像和图形技术的发展,使得人们能够在显微水平下实现对脑结构和脑功能的区域划分,为完善和发展更为细致的脑图谱提供了技术基础。然而现有的大部分高分辨率组织学成像技术均需要先对脑进行切片,该过程中不可避免的组织缺损和形变使其缺乏精确的3维空间信息。为了构建同时具备精确3维空间信息、高分辨率解剖信息以及准确的连接信息的脑图谱,通常研究者们采用磁共振影像和高分辨率组织学影像进行融合的方法,为研究宏观特征的介观机制以及介观特征的宏观表征提供了重要途径。
国内外多个研究团队都在持续发展和利用各种成像技术,并结合大数据处理与分析技术,建立包括啮齿类、非人灵长类动物,以及人类的高分辨率的多组学脑图谱,为研究大脑解剖结构、功能机制以及神经系统疾病提供了有力工具。美国Allen脑研究所一直致力于小鼠脑图谱的构建,通过结合高分辨率显微成像技术与宏观脑影像,发布了包含介观连接的3维图谱标准坐标框架(Oh等,2014)。2020年3月,Allen脑研究所Lydia Ng团队在《Cell》上发布最新的跨模态小鼠脑图谱(Wang等,2020),通过双光子断层扫描技术获得1 675只成年小鼠脑的图像,将这些图像迭代配准平均,构建了一个具有10 μm体素分辨率的平均模板,进一步地,研究人员将多模态脑影像数据作为参考,对该模板进行了细分,标记了43个皮层区域及其分层、329个皮层下灰质结构、81条纤维束和8个脑室结构。与上一版相比,该图谱的空间分辨率提高了1 000倍,更容易分辨解剖学细节,该图谱作为一个通用参考框架,可以整合组织学染色、免疫组织化学、转基因、原位杂交和示踪剂投射等多模态数据,且有良好的可视化工具和交互界面。Schilling等人(2019)则通过整合松鼠猴的磁共振与组织学染色脑影像数据,构建了一个松鼠猴多模态脑图谱,磁共振影像提供了立体坐标信息空间,尼氏染色组织学数据提供了细胞构筑分区标签。该图谱所包含的弥散磁共振纤维追踪可与示踪剂注射结果进行比较,同时局部弥散场可与髓鞘染色结果对应。北京大学王征团队在162只2~9岁的健康食蟹猴上收集分辨率为0.5 mm的磁共振结构影像,创建了一个食蟹猴专用的磁共振影像脑图谱模板,通过与传统的组织学染色结果进行比较,验证了磁共振影像模板的准确性(Lyu等,2021)。2020年8月,德国Katrin Amunts团队在《Science》杂志上发表了最新的人类细胞构筑脑图谱(Julich-Brain atlas)(Amunts等,2020),科研人员采集了10个脑标本的MRI图像和24 000多幅脑组织学切片显微成像,通过对图像进行数字化处理、组织学影像校正与分析、3维重建以及配准,最后完成了Julich-Brain图谱的绘制,它主要具有以下特点:1)基于细胞构筑建立了人脑的微观结构分区;2) 覆盖整个人脑,包括大脑皮层和皮质下核团结构;3) 作为概率图谱,考虑了不同个体的脑在标准空间的概率分布;4) 一个动态的、不断更新但时刻能追溯变化的脑图谱;5) 具有灵活的、允许修改的构建流程,模块化的构建框架适合用于该领域其他相关研究;6) Julich-Brain atlas (www.jubrain.fz-juelich.de)作为欧盟人类脑计划一部分,通过EBRAINS神经信息平台,作为开放资源供全球的科研人员使用。
此外,随着组织透明试剂和方法、高效荧光标记和光片显微镜成像技术的发展,组织透明技术可以对完整的器官甚至部分完整的有机体进行3维成像。该类技术没有大量的切片操作,通过将透明化后的组织切片与神经示踪技术(如化学示踪剂、病毒)和荧光显微成像技术结合,只需将少量的组织学图像拼接完成3维重建,然后将图像配准到MRI空间,即可获得准确的脑区结构连接信息,因此该类技术也大力促进了高分辨率多尺度脑图谱融合技术的发展。中国科学院深圳先进技术研究院毕国强团队将组织切片与透明技术结合,将厚切片组织透明,大大减少了切片工作量与重建误差,并通过自主研发的高通量3维荧光成像VISoR技术与半自动化分析方法,实现了猕猴脑影像的快速获取以及微米级分辨率3维解析,得到了两只猕猴的大脑介观连接图谱(Xu等,2021)。斯坦福大学Karl Deisseroth和Michael Zeineh团队近期在《Nature Communications》杂志上发表了多模态图像配准和连通性分析流程(multimodal image registration and connectivity analysis,MIRACL),这种自动化、开放资源将宏观活体成像、组织透明(CLARITY)显微成像与标准Allen atlas参考框架中的连接性图谱集成在一起,这种集成支持基于纤维束和网络的分析,例如跨网络图谱或沿纤维束的组织学特征研究,以及基于病毒投射终端的连通性分析(Goubran等,2019)。进一步地,通过将MIRACL流程应用于小鼠卒中模型,不仅证明了MRI异常与CLARITY组织染色之间具有很强的一致性,而且还发现了与缺血核心相关区域的急性细胞效应;通过量化来自CLARITY病毒注射的投射终端,提出了改进的连接图谱,并将弥散MRI与CLARITY病毒追踪结合实现不同尺度的连接图谱的比较,利用这种多模态融合的方式解释了中风在纤维束水平的组织学改变。
1.1.2 脑图谱特征层面融合
除了直观地基于跨模态脑影像数据的空间信息构建具有分区信息的脑图谱,研究者们还定义了一系列特征来度量不同脑区的结构和功能特性。比如,由功能磁共振影像可以计算功能局部一致性,该指标能描述局部体素的时间序列的相似性;功能连接(function connectivity,FC)则可以度量不同脑区之间的相关性。基于张量模型的弥散张量成像能提供多个量化弥散性的指标,包括部分各向异性值,它能表征水分子各向异性的扩散情况,其值越大,表明组织的各向异性越明显;径向扩散系数则表示垂直于白质纤维方向的扩散情况,能描述髓鞘化的程度等。通过建立不同影像特征之间的融合模型,一方面可以回答结构和功能的关联关系等基础脑科学问题,另一方面基于融合特征可以挖掘脑疾病的生物标志等。
Misic等人基于215例来自HCP的结构和功能磁共振数据,应用Desikan-Killiany图谱作为模板分别构建结构和功能网络,并应用多元回归模型构建结构和功能之间的关系,结果发现结构和功能在初级感觉区和运动区密切相关,但在高级皮层,尤其是默认网络和凸显网络中,结构和功能的耦合降低,这种结构和功能的分层结构即通常所说的栓系假说理论(tethering hypothesis)(Vázquez-Rodríguez等,2019)。在此基础上,Wu等人(2020)进一步回答了解剖连通性对全脑功能激活的预测能力是否也存在这种层级结构。该研究基于Brainnetome图谱划分的不同亚区构建连接特征,然后建立解剖连接和功能连接两种特征间的预测关系,并比较了这种关系在不同网络中的层级结构以及在不同任务状态下的一致性。结果发现,在初级皮层的预测效果要优于高级皮层,证实了大脑皮层存在着一种共同的层级机制,为解析人类大脑解剖结构和功能激活的关系提供了重要证据。此外,Luo等人(2020)进一步在包含15 000例被试的大数据中,采用独立成分分解的方法分别构建结构网络和功能网络,通过对比结构网络和功能网络的空间一致性在全脑不同区域的分布,同样发现了结构和功能的空间一致性也存在从初级皮层到高级皮层依次降低的层级结构。
在栓系假说的基础上,研究者们进一步提出,这些未受约束的区域对精神疾病的潜在脆弱性,很可能是导致产生精神疾病的原因(van den Heuvel等,2019),因此通过挖掘脑结构和功能的共损伤可能是发现脑疾病的稳定生物标志的一种重要途径。Sui等人(2018)在精神分裂症人群里中通过融合灰质体积、结构连接各向异性分数以及功能低频振幅3种模态发现,凸显网络、胼胝体、中央执行网络和默认网络能作为精神分裂症广义认知损伤的模态特异性生物标记。其中,功能低频振幅特征对认知领域的差异更为敏感,而凸显网络的灰质体积变化和胼胝体的各向异性分数对多个认知领域具有高度一致性和预测性。上海科技大学沈定刚团队则长期致力于将多任务学习模型应用于结构和功能融合来寻找阿尔兹海默症的生物标志(Zhang等,2012),这种方法的基本思想是以最小化疾病分类的精度来作为目标函数,优化多模态的特征选择。在此基础上,为了不断完善模态间的相关性又保持各模态的独立性,引入了各模态的独立性约束(Jie等,2015)和人群间的相似性作为先验知识(Hao等,2020)等。同时,由于不同的脑图谱强调的特征存在差异,因此融合多个脑图谱往往能得到相较于单个脑图谱更好的分类结果,于是进一步提出基于多模板学习思想的融合框架用于阿尔兹海默症的分类研究(Liu等,2016;Yao等,2021a)。此外,Cao等人(2015)则将多视角子空间聚类方法应用到跨模态脑影像特征融合,并用于挖掘阿尔兹海默症的生物标志。美国印第安纳大学的Sporns等人提出嵌入向量表征方法来构建结构连接和功能连接之间的关系,并将该方法应用于面孔识别能力损失患者的结构网络和功能连接模式的相互预测(Rosenthal等,2018)。
1.2 跨模态数据融合
基因组、转录组和蛋白质组等多组学数据以及神经电生理、认知和行为等数据缺乏空间信息,脑图谱为该类型数据提供重要的空间定位信息。因此,基于脑图谱的位置约束,借助先进的机器学习模型实现脑图谱与该类数据的跨模态数据融合,不仅促进了对脑认知和行为的脑影像基础和生物学基础的认识,也辅助理解了底层模态的生物过程是如何影响或改变宏观脑区的结构,最终导致大脑的失调、认知功能损伤和病变。
1.2.1 脑图谱与多组学数据融合
脱氧核糖核酸(deoxyribo nucleic acid,DNA)是生物体发育和正常运作必不可少的生物大分子,DNA中的核苷酸碱基排列顺序构成了遗传信息,该遗传信息可以通过转录过程形成核糖核酸(ribonucleic acid,RNA),其中的信使RNA(message RNA, mRNA)通过翻译产生多肽,形成蛋白质。由遗传信息合成功能性基因产物的过程也就是基因表达过程,由于功能性基因产物的丰度存在差异化,于是产生了多种多样的细胞类型,在不同的生命活动中发挥重要功能。利用脑图谱位置约束,可以关联不同人群遗传信息的差异、定量不同脑区在不同水平的基因表达,从而更直观地构建宏观脑影像表型与多组学信息之间的联系,揭示了脑网络组织层次的潜在分子机制,极大地促进了人们对脑功能的认识,对开发新型神经疾病治疗手段具有重要意义。接下来将从全基因组和基因表达(转录组和蛋白质组)两方面总结脑图谱与多组学信息融合的最新研究成果。
1) 脑图谱与全基因组数据融合。随着测序技术的迅猛发展,全基因组测序成本一降再降,使得研究者们可以对超大规模人群队列展开研究,探索脑结构和功能的遗传机制。基于该类测序结果,一方面可以开展全基因组关联分析(genome-wide association studies,GWAS),即对多个个体在全基因组范围数以百万的单核苷酸多态性(single nucleotide polymorphism,SNP)进行检测获得基因型,进而将基因型与可观测的表型(脑影像等)进行群体水平的统计学分析,根据统计量或显著性p值筛选出最有可能影响该性状的遗传变异(标记)。2020年,澳大利亚昆士兰大学Sarah E. Medland、美国南加州大学Paul M. Thompson和美国北卡教堂山大学分校Jason L. Stein带领的团队联合在《Science》上发文,通过对51 665名个体的脑磁共振成像数据进行全基因组关联荟萃分析,分析了整个皮质和34个已知功能区的表面积和平均厚度,确定了199个重要的位点,并发现其在与产前皮质发育相关的通路中显著富集(Grasby等,2020)。美国北卡罗莱纳大学教堂山分校朱宏图团队则进一步探讨了白质微结构的遗传机理,通过对约4.4万人的大脑扩散磁共振成像数据进行GWAS分析,重复发现了42个先前曾被报道的与白质微结构相关的遗传区域,还额外找到了109个全新的遗传区域,这151个遗传区域能揭示大脑白质微结构遗传性的32.3% (Zhao等,2021)。除了正常人群的GWAS分析,精神病基因组联盟(psychiatric genomic consortium,PGC)则致力于针对不同精神疾病发布大样本的全基因组分析结果。该机构首先于2014年在《Nature》上发布了一项基于36 989例精神分裂症和113 075例正常对照的GWAS分析结果,发现了108个与精神分裂症显著关联的变异位点,其中83个是首次发现,且这些位点主要富集在钙信号通路(Schizophrenia Working Group of the Psychiatric Genomics,2014)。随后,陆续发布了抑郁症(Wray等,2018)、自闭症(Grove等,2019)和双相情感障碍(Stahl等,2019)等精神疾病的大样本GWAS分析结果。2017年,上海交通大学Bio-X研究院师咏勇团队在《Nature Genetics》上发表了一项基于36 180例中国汉族人群的精神分裂症GWAS分析结果,通过与PGC的GWAS结果比对,75%的精神分裂症易感位点能在跨种族分析中得到验证,该项研究首次发现了30个易感的基因组区域,其中7个是中国人群特异的,其余23个是跨种族共有的(Li等,2017)。
另一方面,对于数百例的中小型样本,直接进行GWAS分析往往得不到显著的结果,因此首先需要对数百万的SNP位点进行基于先验或算法的预筛,然后再考察预筛后的SNP序列与脑影像的关联性(谭俊等,2018)。Luo等人(2018)基于PGC发布的精神分裂症的GWAS结果对1 000多例被试的SNP数据进行预筛,然后采用数据驱动的方法融合灰质体积和功能低频振幅开展研究,结果发现精神分裂症患者的灰质体积相较于正常人在丘脑、壳核、双侧颞叶和楔叶降低,与之共变的功能低频振幅成分在额叶处也降低,同时与一组强调CSMD1(CUB and Sushi Multiple Domains 1),CNTNAP2(Contactin Associated Protein 2),DCC(DCC Netrin 1 Receptor)和GABBR2(Gamma-Aminobutyric Acid Type B Receptor Subunit 2)等精神分裂症易感基因的成分关联,且与工作记忆能力降低显著相关。与此类似,挪威的研究组则基于PGC发布的欧洲高加索人GWAS结果计算多基因风险分数(polygenic risk score,PRS),然后应用线性回归模型计算PRS与皮层厚度和灰质体积之间的相关性,结果发现,多基因风险分数越高,精神分裂症患者额颞叶皮质越薄,海马亚区体积越小(Alnæs等,2019)。美国杜兰大学王玉平团队和美国宾夕法尼亚大学的Shen Li团队则开发了一系列基于稀疏约束的影像遗传学融合方法,用于从高维的SNP序列中挖掘与某种表型显著相关的遗传序列,如深度典型相关稀疏自编码器模型(Li等,2020b)、组稀疏联合非负矩阵分解模型(Peng等,2022)和稀疏关联分析(Du等,2020a, b),并将其应用于精神分裂症、阿尔兹海默症和帕金森的影像遗传学研究。
2) 跨模态脑图谱与转录组、蛋白质组数据融合。转录组指细胞或组织内基因转录产物的总和,其可以在RNA的水平上刻画不同基因表达的丰度。微量测序技术以及空间测序技术的发展,使得研究者可以在近单细胞水平对大脑特定位置建立详尽的分子图谱,从而进行更精细的细胞类型鉴定,揭示大脑生理功能的分子基础。通常,构建转录图谱和宏观连接图谱间融合框架的方式分为3种类型(Fornito等,2019):(1)区域表达分析,即确定基因表达的区域差异与某些区域属性(如节点度)之间的关联;(2)关联基因表达分析,旨在检查区域之间的转录耦合如何与网络组织的成对属性相关;(3)基因共表达网络分析,旨在研究成对基因区域表达谱的相关性。
2020年,中国科学院生物物理研究所王晓群团队利用单细胞测序技术,对妊娠16—27周的人脑中海马体的30 416个细胞的基因表达水平进行定量分析,利用tSNE(t-distributed stochastic neighbor embedding)方法鉴定了47种细胞亚型并构建了其发育轨迹,深入阐明了海马在动态变化的发育过程以及记忆功能环路的形成的细胞基础和分子机制,并比较了人类与啮齿类动物的海马的进化过程的关键差异(Zhong等,2020)。2021年,美国加利福尼亚大学圣迭戈分校Eran Mukamel团队与艾伦研究所Hongkui Zeng在《Nature》上发文,通过整合单细胞转录组数据与表观组数据,对来自小鼠初级运动皮层的50万个细胞鉴定到56种不同的神经元类型,并利用新开发的多模态计算与统计方法验证了细胞类型的可重复性,确保了分类的保守性,并鉴定到一致的标记基因与调控元件(Yao等,2021b)。美国约翰霍普金斯大学的Andrew E. Jaffe团队利用最新的10x Genomics Visium技术,克服了之前仅保留细胞核内RNA的局限,对人脑背外侧前额叶皮层进行空间转录组测序,构建基因表达的空间图谱,并通过整合神经精神障碍基因集,显示了精神分裂症和自闭症谱系障碍相关基因的差异层富集表达(Maynard等,2021)。此外,除了对感兴趣的区域和样本进行测序来开展融合研究,另外一类重要的方法是基于已有的基因表达图谱构建全脑影像和基因表达之间的关联关系。目前最全面、应用最广泛的人脑基因表达图谱是艾伦人脑图谱(Allen human brain atlas,AHBA),它包括对3 702个不同解剖位置和6个不同大脑中超过20 000个基因的微阵列测量(Hawrylycz等,2012)。美国耶鲁大学John D. Murray团队结合AHBA图谱和结构磁共振影像数据发现,脑网络的解剖学层次与皮层转录变异产生的空间梯度高度一致,并且在非灵长类动物中也发现了类似的结果(Burt等,2018)。电子科技大学的陈华富团队则基于两个独立的抑郁症数据集,利用偏最小二乘方法研究了多模态脑影像形态相似网络(morphometric similarity networks,MSN)与基因表达之间的相关性,结果发现一系列与抑郁症MSN特征差异显著相关的基因,且这些基因主要与小胶质细胞和神经元细胞相关的生物过程相关,辅助揭示抑郁症的神经影像学变异背后的转录组机理(Li等,2021a)。采用类似的方法,英国剑桥大学Edward T Bullmore团队发现,精神分裂症病例与对照组形态相似性差异的皮质图谱在空间上与神经生物学相关的功能注释和通路的加权基因组合的皮质表达相关,这类融合神经影像和转录数据的综合分析提供了对理解脑疾病遗传风险如何影响脑网络结构变化的新见解(Morgan等,2019)。
由于转录后调控和翻译调控的广泛存在,仅仅通过测量RNA很难获取关于生命活动的最直接信息,因此,研究者们开始在蛋白水平刻画基因的表达丰度,即蛋白质组。2019年,Wang等人(2019)完成了人体29种健康组织的深度蛋白质组和转录组丰度图谱绘制工作,通过使用无标记串联质谱测序技术确认了13 640种蛋白,包括37种新发现的蛋白质,通过外显子测序发现的近一万个氨基酸异构体,只有约1/50能够在蛋白水平找到。在此基础上,Jiang等人(2020)使用TMT 10/MS3质谱策略对来自14个正常个体、32种不同组织的201个样本测序,这类标记定量测序方法的准确性和可重复性比传统的非标定量要高,最终得到了人体量化的蛋白质图谱。该研究还发现组织的蛋白质丰度信息可以解释遗传疾病的表型,而从转录组数据中无法获得此信息,但是该研究只对脑皮层取了两个样本,没有覆盖全脑。2020年,Mathias Uhlén和Jan Mulder团队使用编码蛋白质的转录组数据以及抗体染色的方法对人、猪和鼠的大脑10个主要脑区(23个人脑亚区,30个猪脑亚区,以及17个小鼠脑亚区)的基因表达丰度进行测序和分析(Sjöstedt等,2020),确定了区域表达谱,并比较了3个物种基因表达的相似性和差异性。通过转录测序和蛋白质测比较分析发现,大约50%的基因的转录组和蛋白质表达丰度没有明显统计学意义上的正相关关系,甚至有小部分基因在两个表达水平是负相关关系。此外,蛋白质组学的研究同样为精神疾病的研究带来了新的曙光。2020年,来自美国埃默里大学的Nicholas T. Seyfried团队对453例背外侧前额叶皮层蛋白质组样本进行基因共表达网络分析,发现了6个蛋白模块对阿尔兹海默症(Alzheimer's disease,AD)呈强相关性,其中星形胶质细胞糖代谢相关蛋白与AD进展密切相关,且进一步筛选出3个M4模块的蛋白可在脑脊液中作为AD诊断标志物(Johnson等,2020)。来自该校的另外一组通过对376名参与者背外侧前额叶皮质样本的蛋白质组学数据和抑郁症最新的GWAS数据进行了全蛋白质组关联分析,鉴定到19个基因可通过调节其脑蛋白丰度参与抑郁症的发病,其中9个在独立的全蛋白质组关联分析中被重复鉴定到,为抑郁症的进一步机制研究和治疗提供了有希望的新靶点(Wingo等,2021)。上海科技大学的水雯箐团队则利用新的脑组织跨膜蛋白质谱技术,系统绘制了小鼠多脑区跨膜蛋白质组图谱,利用深度学习方法创建了一个GPCR(G-protein-coupled receptor)家族的虚拟谱图库,并构建了120个G蛋白偶联受体与跨膜蛋白在脑组织中可能形成的内源蛋白互作网络,加深了对跨膜蛋白空间分布的理解,为抗抑郁药物提供了新的靶点(Li等,2021b)。尽管现有的蛋白质组测序技术已经逐步成熟,但目前尚缺乏一个包含空间位置信息、重复性好和可以量化分析的脑蛋白质组谱图。在精准取样方法与先进的质谱测序技术相结合的基础上,通过脑影像数据提供精准的3维坐标,在不久的将来可以实现包含空间位置信息、重复性好且可量化分析的量化脑蛋白质组图谱的获取。
1.2.2 脑图谱与神经电生理数据融合
头皮脑电(electroencephalography,EEG)是一种以高时间分辨率著称的神经信号采集技术,它通过电极能实时记录脑细胞群的自发性和节律性活动。但是,其低空间分辨率大大限制了该技术的发展,因此,大量的研究通过融合EEG和MRI影像来获得同时具有高时间分辨率和高空间分辨率的信号,提高脑电信号的源定位精度并辅助挖掘疾病的生物标志等。电子科技大学尧德中团队长期开展脑电和磁共振影像的融合研究,结合个体磁共振头模型,利用深度网络结构,发展了真实头模型下的脑电时空稀疏源成像技术(Bore等,2021);并近一步结合小波和多变量分析,发展了时变的大尺度网络构建分析方法,揭示了多种认知过程特有的动态大尺度网络信息加工结构(Yi等,2021);此外,通过同步脑电、磁共振采集,建立了磁共振网络和电生理以及个体行为上的关联,发现了认知注意和特定的磁共振功能网络间存在密切的关联关系(Li等,2020a)。
除了以脑电为代表的非侵入式神经电生理测量技术,侵入式脑电测量技术也逐渐发展和普及,并用来提供临床医疗中局部脑区的信号检测,如皮层脑电图(electrocorticography,ECoG)和立体3维脑电图(stereoelectroencephalograph,SEEG)等。ECoG记录电极是放置在颅骨下和皮层上进行脑电信号的记录,其一般用于需要做脑部手术的病人进行短时间急性信号记录。SEEG技术则是通过立体定向定位技术,将一组深度电极植入大脑,用来检测脑电活动、癫痫灶以及功能区。利用植入侵入式电极的临床病人开展研究,能挖掘更高时空分辨率的信号,解析更复杂的神经电生理活动。美国斯坦福大学的Josef Parvizi团队将功能磁共振成像(functional MRI,fMRI)和皮层电图(ECoG)相结合,对5名神经外科患者植入电极,通过功能磁共振成像将ECoG电极定位在默认网络、背侧注意网络和额顶网络,结果发现从MRI中计算获得的FC测量值与在同一大脑中单独记录的ECoG信号具有可重复的空间相关性(Kucyi等,2018)。在此基础上,该团队与首都医科大学附属天坛医院的张建国团队合作在《Nature》子刊上发文(Kucyi等,2020),通过融合SEEG和fMRI影像开展研究,从电生理的角度阐述了默认网络、背侧注意网络和凸显网络3大重要网络之间的拮抗作用。该研究先用fMRI影像对默认网络、注意网络和凸显网络进行空间位置划分,然后探讨了这些网络内和网络间采集的SEEG信号与行为的相关性,结果发现在高频段(70—170 HB),3个网络呈现不同的任务诱发电生理活动,背侧注意网络的高频段响应最快,凸显网络次之,默认网络最慢,且背侧注意网络和默认网络之间的时间滞后、负高频段耦合的幅值与更高的持续注意力表现相关,并在清醒休息期间降低。
1.2.3 脑图谱与认知和行为评分融合
脑影像与认知和行为的融合是通过建立脑影像指标与行为表征之间的映射关系,进而找到与认知行为有关的影像标记物。以往的研究常用单变量的统计分析方法,在组水平上,对脑影像特征与特定认知评分进行相关性分析,但这种方法忽略了被试之间的个体差异,且不具有泛化能力。随着机器学习方法的发展,越来越多的研究开始构建能够使用影像特征在个体水平预测被试认知行为的模型,如连接预测模型(connectome-based predictive modeling,CPM)(Shen等,2017)等。该类方法通常先基于脑图谱来构建影像连接矩阵,然后在训练集中通过机器学习方法来构建影像特征与认知行为之间的预测模型,最后将该模型应用到测试集中来对新的被试的认知行为进行估计。这些基于脑影像特征的个体化预测模型可以用于寻找稳健可靠的影像标记物,并将其应用于实践,辅助临床制定个性化的决策,从而达到精准医疗的目的。
中国科学院自动化研究所脑网络组研究中心与英国伦敦国王学院Gunter Schumann团队合作,基于大样本的青少年纵向数据和机器学习技术,找到了预测青少年抑制控制能力个体化发育的早期神经预测因子(Wang等,2021)。利用数据驱动的方法,发现14岁腹侧注意力网络与皮下核团之间的功能连接可以在个体水平预测5年后抑制控制能力的变化,且预测模型中所识别的功能连接特征还与未来5年内的药物滥用情况有关。此外,该团队基于Brainnetome图谱计算全脑功能连接特征,结合先进的特征选择及稀疏回归的机器学习方法,对360名大学生的4种气质评分进行了个体化水平预测和多中心验证,发现对气质评分具有预测性能的脑区主要集中在前额叶皮层、海马、杏仁核、基底神经节和扣带回等,且4种气质个性指标表现出相似的脑功能模式(Jiang等,2018)。美国耶鲁大学Marvin M Chun团队基于Shen等人(2013)的图谱计算了被试在执行持久注意力时的全脑功能连接矩阵,并基于CPM模型构建了在成人个体水平预测被试的持久注意力的模型(Rosenberg等,2016),该模型能稳定泛化到一批独立的儿童青少年数据中,并成功预测注意力缺陷多动障碍患者的临床注意缺陷。同样基于Shen等人(2013)的图谱,美国哈佛大学的研究团队使用被试在执行经典的发散性思维任务下的fMRI数据计算了功能连接,并找到了与创新性思维有关的功能连接,包括默认、凸显和执行系统中的额叶和顶叶区域(Beaty等,2018)。使用CPM模型和留一交叉验证方法,作者发现基于功能连接的模型能可靠地预测被试的创新能力,并在多个独立数据集中得到泛化。
此外,通过构建影像和行为评分之间的融合关系,对于脑疾病的诊疗也具有非常重要的意义。中国科学院自动化研究所脑网络组研究中心通过与国内近10家精神科医院合作,收集了上千例精神分裂症的多模态脑影像、临床与认知评估等多水平多组学数据,利用机器学习技术,首次发现了纹状体环路功能异常指标能够作为精神分裂症诊断与疗效预测的生物标记。该研究从生物标记的稳定性与可泛化性、个体化精准诊断与疗效预测模型及生物机制解析等方面进行了系统的阐述,并于2020年发表在《Nature Medicine》。另外,该团队通过将静息状态功能MRI和3个临床特征相结合,并利用机器学习的方法构建了预测单个意识障碍患者一年后意识水平的模型(Song等,2018)。该模型在来自两个医疗中心的3个数据集上以大约88%的准确率区分了一年后会恢复意识的患者和不会恢复意识的患者,且还能够确定不同预测因素的预后重要性。韩国Sungkyunkwan大学的Choong-Wan Woo团队和美国达特茅斯学院的Tor D. Wager团队近期在《Nature Medicine》杂志发表了一项关于疼痛神经影像学生物标记的研究,该研究基于Brainnetome图谱构建的全脑功能连接发现了一组稳健的生物标志,可以预测实验诱导的持续性疼痛程度,并在6项研究中重复验证了该信号对临床疼痛的敏感性、特异性和适用性(Lee等,2021)。美国国家精神卫生研究院Alex Martin团队使用静息态功能连接和机器学习方法,构建了基于基线功能连接特征来预测自闭症患者两年后社交自闭症状和适应性功能变化的预测模型(Plitt等,2015)。结果发现,凸显网络、默认网络和额顶任务控制网络相关的连接在预测未来的社交自闭症状和适应性功能中起关键作用。
2 跨模态脑图谱代表性公开数据库
为了促进脑科学领域的发展,国内外研究者们收集并发布了多个大样本公开数据集,本节将重点介绍部分有代表性的跨模态脑图谱公开数据集,如表 1所示。
表 1
跨模态脑图谱代表性公开数据库
Table 1
Representative brain cross-modality public dataset
| 数据集 | 国别 | 总样本 | 年龄/岁 | 物种 | 模态 |
| UK Biobank | 英国 | >40 000 | 40~69 | 健康成人 | sMRI、rfMRI、tfMRI、DWI、基因 |
| HCP | 美国 | 1 200 | 22~35 | 健康成人 | sMRI、rfMRI、tfMRI、HARDI、MEG、基因 |
| CHIMGEN | 中国 | 7 000 | 18~30 | 健康成人 | sMRI、rfMRI、DWI、环境、基因、行为 |
| ABCD | 美国 | 12 000 | 7~10 | 儿童 | sMRI、rfMRI、tfMRI、DWI、基因 |
| PNC | 美国 | 9 498 | 8~21 | 儿童和青少年 | sMRI、rfMRI、DWI、pCASL、基因 |
| IMAGEN | 欧洲 | 2 000 | 14, 16, 19, 22 | 青少年 | sMRI、rfMRI、tfMRI、基因、行为 |
| PING | 美国 | 1 493 | 3~21 | 儿童和青少年 | sMRI、rfMRI、HARDI、基因、认知 |
| ABIDE | 美国 | 1 112 | 5~24 | 自闭症和正常对照 | sMRI、rfMRI、量表 |
| ADNI | 美国 | 1 650 | 50~100 | EMCI、MCI、LMCI、AD和正常对照 | sMRI、rfMRI、PET、基因、CSF |
| ADHD | 美国 | 776 | 7~21 | 注意缺陷多动障碍和正常对照 | sMRI、rfMRI、DWI、量表 |
| PRIME-DE | 美国 | 592 | 0.8~4.5 | 猕猴 | sMRI、fMRI、DWI |
| NCBR | 美国 | 55 | 成年 | 黑猩猩 | sMRI、DWI |
| Allen Brain Atlas | 美国 | / | / | 人、小鼠、猕猴 | sMRI、DWI、荧光显微成像、转录组、免疫组化、细胞类型神经电生理 |
| EBRAINS Reference Atlases | 欧盟 | / | / | 人、大鼠、小鼠 | 细胞构筑、MRI |
| 注:“/”表示无统计信息。 | |||||
1) UK Biobank。是一项针对英国各地50万人群的大规模前瞻性研究(https://www.ukbiobank.ac.uk/),主要目的是挖掘疾病发生前的生物标记,基线招募的参与者年龄在40~69岁之间,收集的模态信息包括结构磁共振(structural MRI,sMRI)、静息态功能磁共振(resting fMRI,rfMRI)、任务态功能磁共振(task fMRI,tfMRI)、弥散加权成像(diffusion weighted imaging,DWI)和基因,是目前世界上除荟萃分析(meta analysis)以外最大的健康人群样本。
2) HCP。人脑连接组计划(Van Essen等,2013)(https://www.humanconnectome.org/)是由美国国立卫生研究院(National Institute of Health, NIH)于2009年出资启动的大规模收集和共享人脑详尽数据的研究计划,其核心目标是采用多模态神经影像技术在脑区水平上描绘人脑结构和功能连接模式、探索个体间的差异以及研究脑结构和功能与行为之间的关系。该数据集的被试为22~35岁年轻健康的成年双胞胎和非双胞胎兄弟姐妹,采集的数据类型包括sMRI、rfMRI、tfMRI、高角分辨率扩散成像(high-angular-resolution diffusion imaging,HARDI)和行为,部分被试还采集了静息态和任务态脑磁图(magnetoencephalography,MEG)。此外,该项目还启动了用于研究人脑毕生变化研究的多个数据采集计划(Harms等,2018;Howell等,2019;Makropoulos等,2018;Somerville等,2018)。
3) CHIMGEN(Chinese imaging genetics)。收集了7 000余名年龄在18~30岁之间的中国汉族健康样本的sMRI、rfMRI、DWI、基因、环境和行为学数据(http://chimgen.tmu.edu.cn/)(Xu等,2020),旨在研究与神经影像和行为表型相关的遗传和环境因素及其相互作用。除微环境变量外,该项目还根据每位受试者从出生到现在的位置,利用卫星遥感和国家统计局数据库收集了数百种定量宏观环境变量,这将有助于发现与神经影像表型有关的新环境因素。
4) ABCD(adolescent brain cognitive development)。青少年脑认知发育数据集(https://abcdstudy.org/),是美国最大的关注青少年大脑发育的纵向数据集。该数据集拟在美国21个站点采集超过10 000名9~10岁青少年的大脑发育数据,以研究大脑结构和功能随年龄发育的变化。数据类型包括sMRI、rfMRI、tfMRI、DWI、基因和多种健康相关的评估量表。
5) PNC(Philadelphia Neurodevelopmental Cohort)。美国费城神经发育队列是一项旨在描绘正常大脑的发育模式和异常模式如何与认知功能障碍和精神症状相关,并探索遗传学如何影响大脑和行为发育以及精神症状的大型儿童队列研究(https://www.med.upenn.edu/bbl/philadelphianeurodevelopmentalcohort.html)。该数据集包括9 498名8~21岁儿童的信息,这些儿童接受了详细的神经精神病学评估。采集的大脑影像包括sMRI、rfMRI、DWI、伪连续动脉自旋标记序列以及遗传信息,目前约有1 000多名被试的多模态的神经影像数据可以通过基因型和表型数据库(dbGaP)下载。
6) IMAGEN。欧洲第1个多中心影像遗传学研究项目(https://imagen-europe.com/)(Schumann等,2010),旨在确定冲动、奖赏和情绪的个体差异的遗传和神经生物学基础,并确定它们对常见精神疾病发展的预测价值。该数据集征募了来自法国、英国、爱尔兰和德国的8个采集中心的2 000+名14岁青少年被试,采集了行为和认知信息、3T脑影像数据和基因数据,并分别在16岁(仅行为数据)、19岁(脑影像和行为数据)和22岁(脑影像和行为数据)时对他们进行了纵向跟踪。脑影像数据包括sMRI、rfMRI以及3种任务状态下的tfMRI。
7) PING(Pediatric Imaging, Neurocognition, and Genetics)。多站点儿科成像、神经认知和遗传学研究队列(http://pingstudy.ucsd.edu/)的主要目标是创建一个大型的行为和成像表型标准化测量资料库。这项横断面研究收集了年龄范围为3~21岁的1 493名儿童的神经影像和基因组数据,包括sMRI、rfMRI、HARDI和基因。该项目评估了参与者包括药物滥用在内的医疗和神经精神疾病的家族史,以及参与者执行功能、注意力、情景记忆、工作记忆、语言和处理速度等信息。
8) ABIDE(autism brain imaging data exchang)。旨在加速对自闭症的理解(https://fcon_1000.pro-jects.nitrc.org/indi/abide/),该数据集由16个国际影像站点合作,公开分享来自539名自闭症患者和573名典型对照的多模态数据,包括sMRI、rfMRI和认知量表信息。
9) ADNI(Alzheimer's Disease Neuroimaging Initiative)。由美国国家生物医学成像与生物工程研究所和美国国立卫生衰老研究所发起(http://adni.loni.usc.edu/),旨在验证可用于阿尔兹海默症临床治疗实验的生物标记物。参与采集的人员主要为50~100岁来自美国与加拿大等地区的老年人,该项目分为多个阶段开展,共包含早期轻度认知损伤(early mild cognitive impairment,EMCI)350例、轻度认知损伤(MCI)400例、晚期轻度认知损伤(late MCI,LMCI)150例、阿尔兹海默症(AD)400例以及正常对照350例。采集的数据类型包括MRI和正电子发射型计算机断层显像(positron emission computed tomography,PET)、遗传学、认知测试、脑脊液和血液生物标志物等。
10) ADHD-200。致力于通过实施开放数据共享加快对注意缺陷多动障碍(attention deficit and hyperactivity disorder,ADHD)神经基础的理解,该数据集包含来自8个独立成像站点的776名被试的静息态fMRI和解剖数据(http://fcon_1000.projects.nitrc.org/indi/adhd200/),其中285例为年龄在7~21岁的ADHD儿童和青少年,其余491例为正常发育个体。此外还包含表型信息量表:诊断状态、ADHD症状测量、年龄、性别、智商和终生用药情况等。
11) PRIMatE-Data Exchange。灵长类数据交换项目(PRIME-DE)的首要目标是为神经影像学研究创建一个开放的资源库(https://fcon_1000.projects.nitrc.org/indi/indiPRIME.html),以促进非人灵长类脑连接组的绘制,该网站整合并共享了来自世界各地实验室的猕猴脑功能、扩散和结构磁共振成像数据集。其中,最新公布了592例包含fMRI、sMRI和DWI的猕猴跨模态脑影像数据,年龄分布为0.8~4.5岁,包括327只雄性和265只雌性。
12) NCBR(National Chimpanzee Brain Resource)。美国国家黑猩猩脑资源中心(http://www.chimpanzeebrain.org/)旨在通过收集和共享黑猩猩神经成像数据和死后脑组织来推动脑科学研究领域的发展。该数据库共包括514只黑猩猩,其中278只黑猩猩在美国Yerkes国家灵长类动物研究中心扫描了磁共振数据,经过丙泊酚麻醉后,在3T MRI扫描仪下获得sMRI和DWI。
13) Allen Brain Institute。美国艾伦脑研究所制作了多个大规模、跨模态的公开数据集(https://portal.brain-map.org/),包括小鼠脑图谱、脊髓图谱、小鼠脑发育图谱、小鼠介观连接图谱、人脑图谱和细胞类型数据库等。小鼠、猕猴和人脑的结构、功能、转录组等多模态数据用于研究脑发育和脑疾病的基因表达。小鼠脑图谱在细胞水平描述了超过21 000个基因的表达;小鼠脑发育图谱提供了4个产前、3个产后时间点的基因表达数据;人脑图谱整合了影像数据和组织学、转录组数据,描述了基因的空间表达信息;小鼠连接图谱采用病毒示踪的方式绘制,旨在帮助科学家了解脑的介观连接方式;基于不断累积的小鼠和人脑的细胞类型数据库,研究人员根据细胞的基因、形态、所在脑区以及神经电生理信号等多模态信息进行细胞分类。
14) EBRAINS。作为欧盟脑计划的基础设施研究(https://ebrains.eu/),集成了人、大鼠和小鼠的脑图谱作为神经科学研究的空间信息参考框架。其中,人脑图谱是德国Katrin Amunts教授领导团队绘制的Julich-Brain Atlas,大鼠和小鼠脑图谱则分别采用了Waxholm Space (WHS) 大鼠脑图谱和美国艾伦脑研究所的小鼠脑图谱CCF第3版。
3 国内外研究进展比较
1) 高质量大数据的获取。高质量大数据的获取是实现跨模态融合的重要前提条件,它的发展离不开高新的成像和采集技术的出现,尽管国内在高新设备的研制、微观脑图谱绘制等核心技术的发展以及实验室的运行体制上,与国际上的优秀团队相比存在较大的差距,但在某些方面已经走在世界前列。中国科学院院士骆清铭团队于2010年研发的显微光学切片断层成像系统(micro-optical sectioning tomography,MOST),在世界上首次获得了小鼠全脑连接图谱,并逐渐发展了全链条“样本处理、3维高分辨全自动成像、大数据处理与可视化”的自主知识产权,居国际领先水平(Li等,2010)。北京大学成功研制出新一代高速高分辨微型化双光子荧光显微镜(Zong等,2021),能获取小鼠在自由行为过程中大脑神经元和神经突触活动清晰、稳定的图像,并且实现了针对同一批神经元长达一个月的追踪记录。同时,该校率先开发出新型的可遗传编码的荧光探针,能够在生理和病理条件下高时空分辨率地检测多巴胺、乙酰胆碱和去甲肾上腺素的释放,相关成果发表在《Cell》和《Science》等学术期刊上。清华大学戴琼海团队研制了“实时超宽场高分辨率成像显微镜”,兼具1 cm×1.2 cm超宽视场、全视场均一的1.2 μm高分辨率、30帧/s高帧率,数据通量高达51亿像素/s,通过对清醒小鼠在体全脑皮层成像等生命科学实验,能实现以宽场高分辨动态成像为基础的脑动态网络结构、神经血管耦合机制和癫痫病理的探索(Fan等,2019)。
在快速发展的高质量大数据采集技术的基础上,开展纵向大队列的数据采集并开放研究者使用等方面,国内的发展与国际相比也存在较大差距(夏明睿和贺永,2018)。尽管国内相关研究团队已经意识到这类问题,并开展了相关工作,如,中国科学院自动化研究所脑网络组研究中心通过与国内近10家精神科医院合作,收集了上千例精神分裂症的多模态脑影像、基因、临床与认知等多水平数据;天津医科大学收集了7 000余例18~30岁的中国汉族健康人群的基因、结构磁共振、扩散张量成像、静息态功能磁共振、环境和行为学数据;北京师范大学发起中国彩巢计划,通过建设一个跨度十年的健康人群脑智毕生(6~85岁)发展队列来勾绘中国人脑毕生发展常模并开展脑与行为的关联机制研究等。但是,在样本量、数据类型等多方面都存在局限性。
2) 高性能计算平台。通过高质量大数据的跨模态融合进行信息挖掘和知识获取,绘制跨模态脑图谱的过程,离不开高性能计算平台提供的海量存储、巨量算力和高效分享技术的支撑。国际上已经启动的国家级脑计划项目中,涉及跨模态脑图谱绘制的任务,无一例外地依赖于高性能计算平台。例如,欧盟脑计划正在建设的EBRAINS是跨国的多中心高性能超算平台,以新建立的欧洲超级计算网络FENIX为硬件载体,该网络由6个领先的来自不同图像(西班牙、法国、意大利、芬兰、瑞士和德国)的高性能计算中心组成。Fenix基础设施的特点是数据存储库和可扩展的超级计算系统紧密相连且集成良好,为研究人员提供扩展的计算资源、交互式计算服务、虚拟机服务和数据存储。目前,Fenix设施拥有超过24.5 PB长期存储空间,17.12 PB的可高速存取的活跃数据存储空间,以及782个节点用于可扩展计算服务,651个节点用于交互式计算服务,224个节点用于虚拟机服务。整个Fenix设施25%的资源分配给欧盟脑计划使用,用以支撑跨模态脑图谱绘制、脑结构和功能建模等研究任务。美国脑计划的细胞普查网络(BRAIN initiative-cell census network,BICCN) 项目的主要数据存放在艾伦脑研究所的服务器上,而原始数据分别存储在3个数据中心,多组学数据在马里兰大学,脑影像数据在匹兹堡超算中心,神经电生理等数据在麻省理工学院的DANDI(distributed archives for neurophysiology data integration)平台上。项目相关的代码工具在GitHub上更新,并依托谷歌Terra云原生计算平台进行定制开发。该项目绘制了哺乳动物初级运动皮层的跨模态图谱,包括细胞类型图谱和介观连接图谱,为更深入地研究哺乳动物大脑其余部分的细胞类型奠定了基础。加拿大的基于网页的CBRAIN(Canadian Brain Imaging Research Platform)平台,是一个托管在GitHub上的开源软件项目,允许神经影像研究人员通过将数据连接到加拿大和世界各地的高性能计算设施来对数据进行计算密集型分析。CBRAIN集成了大量已成为该领域规范的大型神经影像数据集所需的工具,不需要计算机编程技能,也不需要安装任何软件,从而减少了使用这些资源所需的专业知识。日本的“富岳”(Fugaku)超算设施已在2020年6月完成并位居世界第一,拥有7 630 848核,峰值算力达到442 Pflop/s的HPL基准测试分数,服务于日本脑计划。反观国内,目前已建成8个国家级超级计算中心,两台超算系统在世界排名前10位,分别是排名第4的神威太湖之光(93 Pflop/s)和排名第7的天河-2A(61.4 Pflop/s)。这些超算设施多数用于地质、天气和能源等重要国民经济领域,仅有少数用于脑科学的尝试。
3) 先进的融合方法。随着计算机算力和人工智能等技术的发展,国内在跨模态脑图谱融合方法上的确取得了许多重要进展。清华大学戴琼海院士团队与中国科学院生物物理研究所李栋团队合作,综合测评了现有超分辨卷积神经网络模型在显微图像超分辨任务上的表现,提出傅里叶域注意力卷积和傅里叶域注意力生成对抗网络模型,在不同成像条件下实现最优的显微图像超分辨预测和结构光超分辨重建效果,并观测到线粒体内脊、线粒体拟核、内质网和微丝骨架等生物结构的动态互作新行为(Qiao等,2021)。中国科学院自动化研究所作为国内最早开展类脑智能研究的国立研究机构和首个“人工智能学院”牵头承办单位,发展了多种基于统计学、机器学习和深度学习模型的数据融合框架,并应用于宏观脑影像、影像与认知行为以及影像遗传学等多种数据类型的融合,挖掘了一系列脑疾病损伤和认知相关的生物标志。但是,与国际相关优秀团队相比,国内尚处于起步阶段,尤其是在如何充分利用和借鉴图像处理和模式识别领域的新思想、新方法和新技术,将人工智能领域的新进展和跨模态脑图谱数据的特性有机结合,提高现有成像技术的分辨率,实现多尺度数据的信息挖掘以及在重大脑疾病临床中的应用等方面。
4 发展趋势与展望
如何整合不同尺度、不同模态信息构建多模态跨尺度脑图谱,并对其进行生物验证,以及基于全新脑图谱,如何从多层次(从人到动物、从临床到基础)、多模态(神经影像、遗传操作、神经干预和扰动等新技术)的角度解析重大脑疾病的神经机制,形成全新的脑疾病诊疗范式,是跨模态脑图谱数据融合领域国内外共同面对的难题,也是未来的重要发展方向。要实现该重大目标,离不开高质量大数据的获取、巨量的存储和算力以及高效的人工智能算法。
首先,随着对样本量、成像视野和时长,以及时空分辨率的要求日益增高,在获取高质量大数据的同时,成像速度可能成为一个重要的技术瓶颈。突破这一瓶颈的方法之一是打破传统图像处理链条中原始数据采集、图像重建、图像处理和分析之间的界限和时序性,开发新的图像处理方法并运用到数据采集和图像重建中,发展出具体应用导向和面向图像分析的快速高效的成像技术。如何在成像的过程中运用计算前置、稀疏表征、多尺度采样等理念和方法避免时空维度上无意义原始数据的采集,如何将边缘计算、压缩感知等技术和算法应用于在线快速重建,并集成到成像设备中形成成像技术创新,从而在跨模态脑影像处理链条的上游环节提高成像速度和质量,更有利于后续的处理与分析,成为亟待解决的科学问题。此外,为了缩小在大样本队列采集方面与国际的差距,国家也启动了首批2030脑计划,拟逐步系统地建立以发育、老化和重大脑疾病为主的大规模跨模态大数据队列,来弥补国内在这一方面的不足,也进一步为基于跨模态脑图谱大数据开展脑结构和功能的研究奠定基础。
其次,随着高质量成像技术的发展、大样本公开队列的采集和分析需求,多样化的跨模态成像大数据亟需大规模的存储和巨量的算力。近期,德国著名神经科学家Katrin Amunts和Julich超级计算中心主任Thomas Lippert在《Science》上以《大脑研究挑战超级计算》为题撰文,“了解大脑的所有复杂性需要从多个尺度——从基因组学、细胞和突触到整个器官水平的洞察力,这意味着需要处理大量数据”,同时“人脑的复杂程度远超想象,大脑神经回路的天文数字压倒了研究人员,高性能计算的重要性正日益彰显”(Amunts和Lippert,2021)。神经科学中的大数据挑战需要技术解决方案来存储数据(TB到PB范围内),同时通过云提供给更广泛的社区,这在目前的千亿级技术中是不可能的,但可以通过未来的百亿亿级计算能力来实现。处理如此大的数据集会在输入输出级别产生大量计算需求,尽管更高效的输入输出程序和算法正在出现,但计算挑战仍然非常高。因此,随着中国脑计划的陆续启动,海量数据将被采集、汇聚、处理、融合和共享,促进神经科学家与高性能计算领域专家的通力合作,并构建面向脑科学研究的超算平台将成为迫切的需求。
最后,如何构建多元化的模型从多种模态中挖掘出有用的信息是开展跨模态脑图谱融合的最终目的。如何利用多模态融合方法挖掘不同模态间的互补信息,如高时间分辨率和高空间分辨率信息的融合,以及如何融合宏观、介观和微观等不同空间分辨率的信息,构建全新的多尺度的脑图谱并将其应用于脑疾病研究,挖掘稳定的诊疗生物标志将是未来亟待解决的科学问题。随着人工智能技术的发展,将多模态和大样本的高质量图像与人工智能系统、算法的研究进行融合是必然的趋势。因此,结合微(介)观神经元/神经环路尺度以及宏观脑网络尺度,开展多时空脑功能活动信息的融合研究,开发针对性、集成化的平台和工具,不仅能够帮助更好地理解自主生物智能的涌现机制,验证认知神经科学原理,为人工智能的研究提供启发与参考,同时也能为理解脑疾病的发病机理,开发新型预防、诊断和治疗手段提供技术支撑。
总之,尽管国内在跨模态脑图谱数据融合领域取得了很多重要进展,但无论从高质量图像的获取、大规模计算平台的建设,还是跨模态融合算法的构建等方面,相较于国外优秀团队还存在很大的差距。当然,随着国内人工智能和信息科学等领域的迅速发展,国家2030脑计划项目的大力支撑,很多重大挑战和瓶颈问题必将取得重大突破,国内在该领域也必将跻身世界领先位置。
致谢 本文由中国图象图形学学会脑图谱专业委员会组织撰写。该专委会更多详情请见链接:http://www.csig.org.cn/detail/2847。
参考文献
-
Alnæs D, Kaufmann T, van der Meer D, Córdova-Palomera A, Rokicki J, Moberget T, Bettella F, Agartz I, Barch D M, Bertolino A, Brandt C L, Cervenka S, Djurovic S, Doan N T, Eisenacher S, Fatouros-Bergman H, Flyckt L, Di Giorgio A, Haatveit B, Jönsson E G, Kirsch P, Lund M J, Meyer-Lindenberg A, Pergola G, Schwarz E, Smeland O B, Quarto T, Zink M, Andreassen O A, Westlye L T, Karolinska Schizophrenia Project Consortium. 2019. Brain heterogeneity in schizophrenia and its association with polygenic risk. JAMA Psychiatry, 76(7): 739-748 [DOI:10.1001/jamapsychiatry.2019.0257]
-
Amunts K, Lippert T. 2021. Brain research challenges supercomputing. Science, 374(6571): 1054-1055 [DOI:10.1126/science.abl8519]
-
Amunts K, Mohlberg H, Bludau S, Zilles K. 2020. Julich-Brain: a 3D probabilistic atlas of the human brain's cytoarchitecture. Science, 369(6506): 988-992 [DOI:10.1126/science.abb4588]
-
Beaty R E, Kenett Y N, Christensen A P, Rosenberg M D, Benedek M, Chen Q L, Fink A, Qiu J, Kwapil T R, Kane M J, Silvia P J. 2018. Robust prediction of individual creative ability from brain functional connectivity. Proceedings of the National Academy of Sciences of the United States of America, 115(5): 1087-1092 [DOI:10.1073/pnas.1713532115]
-
Bore J C, Li P Y, Jiang L, Ayedh W M A, Chen C L, Harmah D J, Yao D Z, Cao Z H, Xu P. 2021. A long short-term memory network for sparse spatiotemporal EEG source imaging. IEEE Transactions on Medical Imaging, 40(12): 3787-3800 [DOI:10.1109/TMI.2021.3097758]
-
Brodmann K. 1909. Vergleichende Lokalisationslehre der Grosshirnrinde (in German). Leipzig: Johann Ambrosius Barth
-
Burt J B, Demirtaş M, Eckner W J, Navejar N M, Ji J L, Martin W J, Bernacchia A, Anticevic A, Murray J D. 2018. Hierarchy of transcriptomic specialization across human cortex captured by structural neuroimaging topography. Nature Neuroscience, 21(9): 1251-1259 [DOI:10.1038/s41593-018-0195-0]
-
Cao X C, Zhang C Q, Fu H Z, Liu S and Zhang H. 2015. Diversity-induced multi-view subspace clustering//Proceedings of 2015 IEEE Conference on Computer Vision and Pattern Recognition. Boston, USA: IEEE: 586-594 [DOI: 10.1109/CVPR.2015.7298657]
-
Du L, Liu F, Liu K F, Yao X H, Risacher S L, Han J W, Guo L, Saykin A J, Shen L and Alzheimer's Disease Neuroimaging Initiative. 2020a. Identifying diagnosis-specific genotype-phenotype associations via joint multitask sparse canonical correlation analysis and classification. Bioinformatics, 36(Supplement1): 371-379 [DOI: 10.1093/bioinformatics/btaa434]
-
Du L, Liu K F, Yao X H, Risacher S L, Han J W, Saykin A J, Guo L, Shen L. 2020b. Detecting genetic associations with brain imaging phenotypes in Alzheimer's disease via a novel structured SCCA approach. Medical Image Analysis, 61: #101656 [DOI:10.1016/j.media.2020.101656]
-
Fan J T, Suo J L, Wu J M, Xie H, Shen Y B, Chen F, Wang G J, Cao L C, Jin G F, He Q S, Li T F, Luan G M, Kong L J, Zheng Z R, Dai Q H. 2019. Video-rate imaging of biological dynamics at centimetre scale and micrometre resolution. Nature Photonics, 13(11): 809-816 [DOI:10.1038/s41566-019-0474-7]
-
Fan L Z, Li H, Zhuo J J, Zhang Y, Wang J J, Chen L F, Yang Z Y, Chu C Y, Xie S M, Laird A R, Fox P T, Eickhoff S B, Yu C S, Jiang T Z. 2016. The human brainnetome atlas: a new brain atlas based on connectional architecture. Cerebral Cortex, 26(8): 3508-3526 [DOI:10.1093/cercor/bhw157]
-
Fornito A, Arnatkevičiūt A, Fulcher B D. 2019. Bridging the gap between connectome and transcriptome. Trends in Cognitive Sciences, 23(1): 34-50 [DOI:10.1016/j.tics.2018.10.005]
-
Glasser M F, Coalson T S, Robinson E C, Hacker C D, Harwell J, Yacoub E, Ugurbil K, Andersson J, Bec
km ann C F, Jenkinson M, Smith S M, Van Essen D C. 2016. A multi-modal parcellation of human cerebral cortex. Nature, 536(7615): 171-178 [DOI:10.1038/nature18933] -
Goubran M, Leuze C, Hsueh B, Aswendt M, Ye L, Tian Q Y, Cheng M Y, Crow A, Steinberg G K, McNab J A, Deisseroth K, Zeineh M. 2019. Multimodal image registration and connectivity analysis for integration of connectomic data from microscopy to MRI. Nature Communications, 10(1): #5504 [DOI:10.1038/s41467-019-13374-0]
-
Grasby K L, Jahanshad N, Painter J N, Colodro-Conde L, Bralten J, Hibar D P, Lind P A, Pizzagalli F, Ching C R K, McMahon M A B, Shatokhina N, Zsembik L C P, Thomopoulos S I, Zhu A H, Strike L T, Agartz I, Alhusaini S, Almeida M A A, Alnæs D, Amlien I K, Andersson M, Ard T, Armstrong N J, Ashley-Koch A, Atkins J R, Bernard M, Brouwer R M, Buimer E E L, Bülow R, Bürger C, Cannon D M, Chakravarty M, Chen Q, Cheung J W, Couvy-Duchesne B, Dale A M, Dalvie S, de Araujo T K, de Zubicaray G I, de Zwarte S M C, den Braber A, Doan N T, Dohm K, Ehrlich S, Engelbrecht H R, Erk S, Fan C C, Fedko I O, Foley S F, Ford J M, Fukunaga M, Garrett M E, Ge T, Giddaluru S, Goldman A L, Green M J, Groenewold N A, Grotegerd D, Gurholt T P, Gutman B A, Hansell N K, Harris M A, Harrison M B, Haswell C C, Hauser M, Herms S, Heslenfeld D J, Ho N F, Hoehn D, Hoffmann P, Holleran L, Hoogman M, Hottenga J J, Ikeda M, Janowitz D, Jansen I E, Jia T Y, Jockwitz C, Kanai R, Karama S, Kasperaviciute D, Kaufmann T, Kelly S, Kikuchi M, Klein M, Knapp M, Knodt A R, Krämer B, Lam M, Lancaster T M, Lee P H, Lett T A, Lewis L B, Lopes-Cendes I, Luciano M, Macciardi F, Marquand A F, Mathias S R, Melzer T R, Milaneschi Y, Mirza-Schreiber N, Moreira J C V, Mühleisen T W, Müller-Myhsok B, Najt P, Nakahara S, Nho K, Olde Loohuis L M, Orfanos D P, Pearson J F, Pitcher T L, Pütz B, Quidé Y, Ragothaman A, Rashid F M, Reay W R, Redlich R, Reinbold C S, Repple J, Richard G, Riedel B C, Risacher S L, Rocha C S, Mota N R, Salminen L, Saremi A, Saykin A J, Schlag F, Schmaal L, Schofield P R, Secolin R, Shapland C Y, Shen L, Shin J, Shumskaya E, Sønderby I E, Sprooten E, Tansey K E, Teumer A, Thalamuthu A, Tordesillas-Gutiérrez D, Turner J A, Uhlmann A, Vallerga C L, van der Meer D, van Donkelaar M M J, van Eijk L, van Erp T G M, van Haren N E M, van Rooij D, van Tol M J, Veldink J H, Verhoef E, Walton E, Wang M Y, Wang Y P, Wardlaw J M, Wen W, Westlye L T, Whelan C D, Witt S H, Wittfeld K, Wolf C, Wolfers T, Wu J Q, Yasuda C L, Zaremba D, Zhang Z, Zwiers M P, Artiges E, Assareh A A, Ayesa-Arriola R, Belger A, Brandt C L, Brown G G, Cichon S, Curran J E, Davies G E, Degenhardt F, Dennis M F, Dietsche B, Djurovic S, Doherty C P, Espiritu R, Garijo D, Gil Y, Gowland P A, Green R C, Häusler A N, Heindel W, Ho B C, Hoffmann W U, Holsboer F, Homuth G, Hosten N, Jack C R Jr, Jang M, Jansen A, Kimbrel N A, Kolskår K, Koops S, Krug A, Lim K O, Luykx J J, Mathalon D H, Mather K A, Mattay V S, Matthews S, Mayoral Van Son J, McEwen S C, Melle I, Morris D W, Mueller B A, Nauck M, Nordvik J E, Nöthen M M, O'Leary D S, Opel N, Martinot M L P, Pike G B, Preda A, Quinlan E B, Rasser P E, Ratnakar V, Reppermund S, Steen V M, Tooney P A, Torres F R, Veltman D J, Voyvodic J T, Whelan R, White T, Yamamori H, Adams H H H, Bis J C, Debette S, Decarli C, Fornage M, Gudnason V, Hofer E, Ikram M A, Launer L, Longstreth W T, Lopez O L, Mazoyer B, Mosley T H, Roshchupkin G V, Satizabal C L, Schmidt R, Seshadri S, Yang Q, Alzheimer's Disease Neuroimaging Initiative, CHARGE Consortium, EPIGEN Consortium, IMAGEN Consortium, SYS Consortium, Parkinson's Progression Markers Initiative, Alvim M K M, Ames D, Anderson T J, Andreassen O A, Arias-Vasquez A, Bastin M E, Baune B T, Beckham J C, Blangero J, Boomsma D I, Brodaty H, Brunner H G, Buckner R L, Buitelaar J K, Bustillo J R, Cahn W, Cairns M J, Calhoun V, Carr V J, Caseras X, Caspers S, Cavalleri G L, Cendes F, Corvin A, Crespo-Facorro B, Dalrymple-Alford J C, Dannlowski U, de Geus E J C, Deary I J, Delanty N, Depondt C, Desrivières S, Donohoe G, Espeseth T, Fernández G, Fisher S E, Flor H, Forstner A J, Francks C, Franke B, Glahn D C, Gollub R L, Grabe H J, Gruber O, Håberg A K, Hariri A R, Hartman C A, Hashimoto R, Heinz A, Henskens F A, Hillegers M H J, Hoekstra P J, Holmes A J, Hong L E, Hopkins W D, Hulshoff Pol H E, Jernigan T L, Jönsson E G, Kahn R S, Kennedy M A, Kircher T T J, Kochunov P, Kwok J B J, Le Hellard S, Loughland C M, Martin N G, Martinot J L, McDonald C, McMahon K L, Meyer-Lindenberg A, Michie P T, Morey R A, Mowry B, Nyberg L, Oosterlaan J, Ophoff R A, Pantelis C, Paus T, Pausova Z, Penninx B, Polderman T J C, Posthuma D, Rietschel M, Roffman J L, Rowland L M, Sachdev P S, Sämann P G, Schall U, Schumann G, Scott R J, Sim K, Sisodiya S M, Smoller J W, Sommer I E, St Pourcain B, Stein D J, Toga A W, Trollor J N, Van der Wee N J A, van't Ent D, Völzke H, Walter H, Weber B, Weinberger D R, Wright M J, Zhou J, Stein J L, Thompson P M, Medland S E, Enhancing NeuroImaging Genetics through Meta-Analysis Consortium-Genetics Working Group. 2020. The genetic architecture of the human cerebral cortex. Science, 367(6484): #6690 [DOI:10.1126/science.aay6690]
-
Grove J, Ripke S, Als T D, Mattheisen M, Walters R K, Won H, Pallesen J, Agerbo E, Andreassen O A, Anney R, Awashti S, Belliveau R, Bettella F, Buxbaum J D, Bybjerg-Grauholm J, Bækvad-Hansen M, Cerrato F, Chambert K, Christensen J H, Churchhouse C, Dellenvall K, Demontis D, De Rubeis S, Devlin B, Djurovic S, Dumont A L, Goldstein J I, Hansen C S, Hauberg M E, Hollegaard M V, Hope S, Howrigan D P, Huang H L, Hultman C M, Klei L, Maller J, Martin J, Martin A R, Moran J L, Nyegaard M, Nærland T, Palmer D S, Palotie A, Pedersen C B, Pedersen M G, Dpoterba T, Poulsen J B, Pourcain B S, Qvist P, Rehnström K, Reichenberg A, Reichert J, Robinson E B, Roeder K, Roussos P, Saemundsen E, Sandin S, Satterstrom F K, Davey Smith G, Stefansson H, Steinberg S, Stevens C R, Sullivan P F, Turley P, Walters G B, Xu X Y, Autism Spectrum Disorder Working Group of the Psychiatric Genomics Consortium, Bupgen, Major Depressive Disorder Working Group of the Psychiatric Genomics Consortium, 23andMe Research Team, Stefansson K, Geschwind D H, Nordentoft M, Hougaard D M, Werge T, Mors O, Mortensen P B, Neale B M, Daly M J, Børglum A D. 2019. Identification of common genetic risk variants for autism spectrum disorder. Nature Genetics, 51(3): 431-444 [DOI:10.1038/s41588-019-0344-8]
-
Hao X K, Bao Y J, Guo Y C, Yu M, Zhang D Q, Risacher S L, Saykin A J, Yao X H, Shen L, Alzheimer's Disease Neuroimaging Initiative. 2020. Multi-modal neuroimaging feature selection with consistent metric constraint for diagnosis of Alzheimer's disease. Medical Image Analysis, 60: #101625 [DOI:10.1016/j.media.2019.101625]
-
Harms M P, Somerville L H, Ances B M, Andersson J, Barch D M, Bastiani M, Bookheimer S Y, Brown T B, Buckner R L, Burgess G C, Coalson T S, Chappell M A, Dapretto M, Douaud G, Fischl B, Glasser M F, Greve D N, Hodge C, Jamison K W, Jbabdi S, Kandala S, Li X F, Mair R W, Mangia S, Marcus D, Mascali D, Moeller S, Nichols T E, Robinson E C, Salat D H, Smith S M, Sotiropoulos S N, Terpstra M, Thomas K M, Tisdall M D, Ugurbil K, van der Kouwe A, Woods R P, Zöllei L, Van Essen D C, Yacoub E. 2018. Extending the human connectome project across ages: imaging protocols for the lifespan development and aging projects. NeuroImage, 183: 972-984 [DOI:10.1016/j.neuroimage.2018.09.060]
-
Hawrylycz M J, Lein E S, Guillozet-Bongaarts A L, Shen E H, Ng L, Miller J A, van de Lagemaat L N, Smith K A, Ebbert A, Riley Z L, Abajian C, Bec
km ann C F, Bernard A, Bertagnolli D, Boe A F, Cartagena P M, Chakravarty M M, Chapin M, Chong J, Dalley R A, Daly B D, Dang C, Datta S, Dee N, Dolbeare T A, Faber V, Feng D, Fowler D R, Goldy J, Gregor B W, Haradon Z, Haynor D R, Hohmann J G, Horvath S, Howard R E, Jeromin A, Jochim J M, Kinnunen M, Lau C, Lazarz E T, Lee C, Lemon T A, Li L, Li Y, Morris J A, Overly C C, Parker P D, Parry S E, Reding M, Royall J J, Schulkin J, Sequeira P A, Slaughterbeck C R, Smith S C, Sodt A J, Sunkin S M, Swanson B E, Vawter M P, Williams D, Wohnoutka P, Zielke H R, Geschwind D H, Hof P R, Smith S M, Koch C, Grant S G N, Jones A R. 2012. An anatomically comprehensive atlas of the adult human brain transcriptome. Nature, 489(7416): 391-399 [DOI:10.1038/nature11405] -
Howell B R, Styner M A, Gao W, Yap P T, Wang L, Baluyot K, Yacoub E, Chen G, Potts T, Salzwedel A, Li G, Gilmore J H, Piven J, Smith J K, Shen D G, Ugurbil K, Zhu H T, Lin W L, Elison J T. 2019. The UNC/UMN baby connectome project (BCP): an overview of the study design and protocol development. NeuroImage, 185: 891-905 [DOI:10.1016/j.neuroimage.2018.03.049]
-
Jiang R T, Calhoun V D, Zuo N M, Lin D D, Li J, Fan L Z, Qi S L, Sun H L, Fu Z N, Song M, Jiang T Z, Sui J. 2018. Connectome-based individualized prediction of temperament trait scores. NeuroImage, 183: 366-374 [DOI:10.1016/j.neuroimage.2018.08.038]
-
Jiang L H, Wang M, Lin S, Aaron E R. 2020. A quantitative proteome map of the human body. Cell, 183(1): 269-283
-
Jie B, Zhang D Q, Cheng B, Shen D G, Alzheimer's Disease Neuroimaging Initiative. 2015. Manifold regularized multitask feature learning for multimodality disease classification. Human Brain Mapping, 36(2): 489-507 [DOI:10.1002/hbm.22642]
-
Johnson E C B, Dammer E B, Duong D M, Ping L Y, Zhou M T, Yin L M, Higginbotham L A, Guajardo A, White B, Troncoso J C, Thambisetty M, Montine T J, Lee E B, Trojanowski J Q, Beach T G, Reiman E M, Haroutunian V, Wang M H, Schadt E, Zhang B, Dickson D W, Ertekin-Taner N, Golde T E, Petyuk V A, De Jager P L, Bennett D A, Wingo T S, Rangaraju S, Hajjar I, Shulman J M, Lah J J, Levey A I, Seyfried N T. 2020. Large-scale proteomic analysis of Alzheimer's disease brain and cerebrospinal fluid reveals early changes in energy metabolism associated with microglia and astrocyte activation. Nature Medicine, 26(5): 769-780 [DOI:10.1038/s41591-020-0815-6]
-
Kucyi A, Daitch A, Raccah O, Zhao B T, Zhang C, Esterman M, Zeineh M, Halpern C H, Zhang K, Zhang J G, Parvizi J. 2020. Electrophysiological dynamics of antagonistic brain networks reflect attentional fluctuations. Nature Communications, 11(1): #325 [DOI:10.1038/s41467-019-14166-2]
-
Kucyi A, Schrouff J, Bickel S, Foster B L, Shine J M, Parvizi J. 2018. Intracranial electrophysiology reveals reproducible intrinsic functional connectivity within human brain networks. The Journal of Neuroscience, 38(17): 4230-4242 [DOI:10.1523/JNEUROSCI.0217-18.2018]
-
Lee J J, Kim H J, Čeko M, Park B Y, Lee S A, Park H, Roy M, Kim S G, Wager T D, Woo C W. 2021. A neuroimaging biomarker for sustained experimental and clinical pain. Nature Medicine, 27(1): 174-182 [DOI:10.1038/s41591-020-1142-7]
-
Li A A, Gong H, Zhang B, Wang Q D, Yan C, Wu J P, Liu Q, Zeng S Q, Luo Q M. 2010. Micro-optical sectioning tomography to obtain a high-resolution atlas of the mouse brain. Science, 330(6009): 1404-1408 [DOI:10.1126/science.1191776]
-
Li F L, Tao Q, Peng W J, Zhang T, Si Y J, Zhang Y S, Yi C L, Biswal B, Yao D Z, Xu P. 2020a. Inter-subject P300 variability relates to the efficiency of brain networks reconfigured from resting-to task-state: evidence from a simultaneous event-related EEG-fMRI study. NeuroImage, 205: #116285 [DOI:10.1016/j.neuroimage.2019.116285]
-
Li G, Han D P, Wang C, Hu W X, Calhoun V D, Wang Y P. 2020b. Application of deep canonically correlated sparse autoencoder for the classification of schizophrenia. Computer Methods and Programs in Biomedicine, 183: #105073 [DOI:10.1016/j.cmpb.2019.105073]
-
Li J, Seidlitz J, Suckling J, Fan F Y, Ji G J, Meng Y, Yang S Q, Wang K, Qiu J, Chen H F, Liao W. 2021a. Cortical structural differences in major depressive disorder correlate with cell type-specific transcriptional signatures. Nature Communications, 12(1): #1647 [DOI:10.1038/s41467-021-21943-5]
-
Li S S, Luo H Q, Lou R H, Tian C P, Miao C, Xia L S, Pan C, Duan X X, Dang T, Li H, Fan C Y, Tang P, Zhang Z Z, Liu Y, Li Y X, Xu F, Zhang Y Y, Zhong G S, Hu J, Shui W Q. 2021b. Multiregional profiling of the brain transmembrane proteome uncovers novel regulators of depression. Science Advances, 7(30): #0634 [DOI:10.1126/sciadv.abf0634]
-
Li Z Q, Chen J H, Yu H, He L, Xu Y F, Zhang D, Yi Q Z, Li C G, Li X W, Shen J W, Song Z J, Ji W D, Wang M, Zhou J, Chen B Y, Liu Y H, Wang J Q, Wang P, Yang P, Wang Q Z, Feng G Y, Liu B X, Sun W S, Li B J, He G, Li W D, Wan C L, Xu Q, Li W J, Wen Z J, Liu K, Huang F, Ji J, Ripke S, Yue W H, Sullivan P F, O'Donovan M C, Shi Y Y. 2017. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics, 49(11): 1576-1583 [DOI:10.1038/ng.3973]
-
Liu M X, Zhang D Q, Shen D G. 2016. Relationship induced multi-template learning for diagnosis of Alzheimer's disease and mild cognitive impairment. IEEE Transactions on Medical Imaging, 35(6): 1463-1474 [DOI:10.1109/TMI.2016.2515021]
-
Luo N, Sui J, Abrol A, Chen J Y, Turner J A, Damaraju E, Fu Z N, Fan L Z, Lin D D, Zhuo C J, Xu Y, Glahn D C, Rodrigue A L, Banich M T, Pearlson G D, Calhoun V D. 2020. Structural brain architectures match intrinsic functional networks and vary across domains: a study from 15 000+ individuals. Cerebral Cortex, 30(10): 5460-5470 [DOI:10.1093/cercor/bhaa127]
-
Luo N, Sui J, Chen J Y, Zhang F Q, Tian L, Lin D D, Song M, Calhoun V D, Cui Y, Vergara V M, Zheng F F, Liu J Y, Yang Z Y, Zuo N M, Fan L Z, Xu K B, Liu S F, Li J, Xu Y, Liu S, Lv L X, Chen J, Chen Y C, Guo H, Li P, Lu L, Wan P, Wang H N, Wang H L, Yan H, Yan J, Yang Y F, Zhang H X, Zhang D, Jiang T Z. 2018. A schizophrenia-related genetic-brain-cognition pathway revealed in a large Chinese population. eBioMedicine, 37: 471-482 [DOI:10.1016/j.ebiom.2018.10.009]
-
Lyu Q M, Yan M C, Shen X Y, Wu J, Yu W W, Yan S Y, Yang F, Zeljic K, Shi Y Q, Zhou Z F, Lv L B, Hu X T, Menon R, Wang Z. 2021. Normative analysis of individual brain differences based on a population MRI-based atlas of cynomolgus macaques. Cerebral Cortex, 31(1): 341-355 [DOI:10.1093/cercor/bhaa229]
-
Makropoulos A, Robinson E C, Schuh A, Wright R, Fitzgibbon S, Bozek J, Counsell S J, Steinweg J, Vecchiato K, Passerat-Palmbach J, Lenz G, Mortari F, Tenev T, Duff E P, Bastiani M, Cordero-Grande L, Hughes E, Tusor N, Tournier J D, Hutter J, Price A N, Teixeira R P A G, Murgasova M, Victor S, Kelly C, Rutherford M A, Smith S M, Edwards A D, Hajnal J V, Jenkinson M, Rueckert D. 2018. The developing human connectome project: a minimal processing pipeline for neonatal cortical surface reconstruction. NeuroImage, 173: 88-112 [DOI:10.1016/j.neuroimage.2018.01.054]
-
Maynard K R, Collado-Torres L, Weber L M, Uytingco C, Barry B K, Williams S R, Catallini J L II, Tran M N, Besich Z, Tippani M, Chew J, Yin Y F, Kleinman J E, Hyde T M, Rao N, Hicks S C, Martinowich K, Jaffe A E. 2021. Transcriptome-scale spatial gene expression in the human dorsolateral prefrontal cortex. Nature Neuroscience, 24(3): 425-436 [DOI:10.1038/s41593-020-00787-0]
-
Morgan S E, Seidlitz J, Whitaker K J, Romero-Garcia R, Clifton N E, Scarpazza C, van Amelsvoort T, Marcelis M, van Os J, Donohoe G, Mothersill D, Corvin A, Pocklington A, Raznahan A, McGuire P, Vértes P E, Bullmore E T. 2019. Cortical patterning of abnormal morphometric similarity in psychosis is associated with brain expression of schizophrenia-related genes. Proceedings of the National Academy of Sciences of the United States of America, 116(19): 9604-9609 [DOI:10.1073/pnas.1820754116]
-
Oh S W, Harris J A, Ng L, Winslow B, Cain N, Mihalas S, Wang Q X, Lau C, Kuan L, Henry A M, Mortrud M T, Ouellette B, Nguyen T N, Sorensen S A, Slaughterbeck C R, Wakeman W, Li Y, Feng D, Ho A, Nicholas E, Hirokawa K E, Bohn P, Joines K M, Peng H C, Hawrylycz M J, Phillips J W, Hohmann J G, Wohnoutka P, Gerfen C R, Koch C, Bernard A, Dang C, Jones A R, Zeng H K. 2014. A mesoscale connectome of the mouse brain. Nature, 508(7495): 207-214 [DOI:10.1038/nature13186]
-
Peng P, Zhang Y P, Ju Y F, Wang K M, Li G, Vince D C, Wang Y P. 2022. Group sparse joint non-negative matrix factorization on orthogonal subspace for multi-modal imaging genetics data analysis. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 19(1): 479-490 [DOI:10.1109/TCBB.2020.2999397]
-
Plitt M, Barnes K A, Wallace G L, Kenworthy L, Martin A. 2015. Resting-state functional connectivity predicts longitudinal change in autistic traits and adaptive functioning in autism. Proceedings of the National Academy of Sciences of the United States of America, 112(48): E6699-E6706 [DOI:10.1073/pnas.1510098112]
-
Qiao C, Li D, Guo Y T, Liu C, Jiang T, Dai Q H, Li D. 2021. Evaluation and development of deep neural networks for image super-resolution in optical microscopy. Nature Methods, 18(2): 194-202 [DOI:10.1038/s41592-020-01048-5]
-
Rosenberg M D, Finn E S, Scheinost D, Papademetris X, Shen X L, Constable R T, Chun M M. 2016. A neuromarker of sustained attention from whole-brain functional connectivity. Nature Neuroscience, 19(1): 165-171 [DOI:10.1038/nn.4179]
-
Rosenthal G, Váša F, Griffa A, Hagmann P, Amico E, Goñi J, Avidan G, Sporns O. 2018. Mapping higher-order relations between brain structure and function with embedded vector representations of connectomes. Nature Communications, 9(1): #2178 [DOI:10.1038/s41467-018-04614-w]
-
Schilling K G, Gao Y R, Christian M, Janve V, Stepniewska I, Landman B A, Anderson A W. 2019. A web-based atlas combining MRI and histology of the squirrel monkey brain. Neuroinformatics, 17(1): 131-145 [DOI:10.1007/s12021-018-9391-z]
-
Schizophrenia Working Group of the Psychiatric Genomics Consortium. 2014. Biological insights from 108 schizophrenia-associated genetic loci. Nature, 511(7510): 421-427 [DOI:10.1038/nature13595]
-
Schumann G, Loth E, Banaschewski T, Barbot A, Barker G, Büchel C, Conrod P J, Dalley J W, Flor H, Gallinat J, Gallinat J, Garavan H, Heinz A, Itterman B, Lathrop M, Mallik C, Mann K, Martinot J L, Paus T, Poline J B, Robbins T W, Rietschel M, Reed L, Smolka M, Spanagel R, Speiser C, Stephens D N, Ströhle A, Struve M. 2010. The IMAGEN study: reinforcement-related behaviour in normal brain function and psychopathology. Molecular Psychiatry, 15(12): 1128-1139 [DOI:10.1038/mp.2010.4]
-
Shen X, Tokoglu F, Papademetris X, Constable R T. 2013. Groupwise whole-brain parcellation from resting-state fMRI data for network node identification. Neuroimage, 82: 403-415
-
Shen X L, Finn E S, Scheinost D, Rosenberg M D, Chun M M, Papademetris X, Constable R T. 2017. Using connectome-based predictive modeling to predict individual behavior from brain connectivity. Nature Protocols, 12(3): 506-518 [DOI:10.1038/nprot.2016.178]
-
Sjöstedt E, Zhong W, Fagerberg L, Karlsson M, Mitsios N, Adori C, Oksvold P, Edfors F, Limiszewska A, Hikmet F, Huang J R, Du Y T, Lin L, Dong Z Y, Yang L, Liu X, Jiang H, Xu X, Wang J, Yang H M, Bolund L, Mardinoglu A, Zhang C, von Feilitzen K, Lindskog C, Pontén F, Luo Y L, Hökfelt T, Uhlén M, Mulder J. 2020. An atlas of the protein-coding genes in the human, pig, and mouse brain. Science, 367(6482): #5947 [DOI:10.1126/science.aay5947]
-
Somerville L H, Bookheimer S Y, Buckner R L, Burgess G C, Curtiss S W, Dapretto M, Elam J S, Gaffrey M S, Harms M P, Hodge C, Kandala S, Kastman E K, Nichols T E, Schlaggar B L, Smith S M, Thomas K M, Yacoub E, Van Essen D C, Barch D M. 2018. The lifespan human connectome project in development: a large-scale study of brain connectivity development in 5-21 year olds. NeuroImage, 183: 456-468 [DOI:10.1016/j.neuroimage.2018.08.050]
-
Song M, Yang Y, He J H, Yang Z Y, Yu S, Xie Q Y, Xia X Y, Dang Y Y, Zhang Q, Wu X H, Cui Y, Hou B, Yu R H, Xu R X, Jiang T Z. 2018. Prognostication of chronic disorders of consciousness using brain functional networks and clinical characteristics. eLife, 7: #36173 [DOI:10.7554/eLife.36173]
-
Stahl E A, Breen G, Forstner A J, McQuillin A, Ripke S, Trubetskoy V, Mattheisen M, Wang Y P, Coleman J R I, Gaspar H A, de Leeuw C A, Steinberg S, Pavlides J M W, Trzaskowski M, Byrne E M, Pers T H, Holmans P A, Richards A L, Abbott L, Agerbo E, Akil H, Albani D, Alliey-Rodriguez N, Als T D, Anjorin A, Antilla V, Awasthi S, Badner J A, Bøkvad-Hansen M, Barchas J D, Bass N, Bauer M, Belliveau R, Bergen S E, Pedersen C B, Bæen E, Boks M P, Boocock J, Budde M, Bunney W, Burmeister M, Bybjerg-Grauholm J, Byerley W, Casas M, Cerrato F, Cervantes P, Chambert K, Charney A W, Chen D F, Churchhouse C, Clarke T K, Coryell W, Craig D W, Cruceanu C, Curtis D, Czerski P M, Dale A M, de Jong S, Degenhardt F, Del-Favero J, DePaulo J R, Djurovic S, Dobbyn A L, Dumont A, Elvsåshagen T, Escott-Price V, Fan C C, Fischer S B, Flickinger M, Foroud T M, Forty L, Frank J, Fraser C, Freimer N B, Frisén L, Gade K, Gage D, Garnham J, Giambartolomei C, Pedersen M G, Goldstein J, Gordon S D, Gordon-Smith K, Green E K, Green M J, Greenwood T A, Grove J, Guan W H, Guzman-Parra J, Hamshere M L, Hautzinger M, Heilbronner U, Herms S, Hipolito M, Hoffmann P, Holland D, Huckins L, Jamain S, Johnson J S, Juréus A, Kandaswamy R, Karlsson R, Kennedy J L, Kittel-Schneider S, Knowles J A, Kogevinas M, Koller A C, Kupka R, Lavebratt C, Lawrence J, Lawson W B, Leber M, Lee P H, Levy S E, Li J Z, Liu C Y, Lucae S, Maaser A, MacIntyre D J, Mahon P B, Maier W, Martinsson L, McCarroll S, McGuffin P, McInnis M G, McKay J D, Medeiros H, Medland S E, Meng F, Milani L, Montgomery G W, Morris D W, Mühleisen T W, Mullins N, Nguyen H, Nievergelt C M, Adolfsson A N, Nwulia E A, O'Donovan C, Loohuis L M O, Ori A P S, Oruc L, Ösby U, Perlis R H, Perry A, Pfennig A, Potash J B, Purcell S M, Regeer E J, Reif A, Reinbold C S, Rice J P, Rivas F, Rivera M, Roussos P, Ruderfer D M, Ryu E, Sánchez-Mora C, Schatzberg A F, Scheftner W A, Schork N J, Shannon Weickert C, Shehktman T, Shilling P D, Sigurdsson E, Slaney C, Smeland O B, Sobell J L, Søholm Hansen C, Spijker A T, St Clair D, Steffens M, Strauss J S, Streit F, Strohmaier J, Szelinger S, Thompson R C, Thorgeirsson T E, Treutlein J, Vedder H, Wang W Q, Watson S J, Weickert T W, Witt S H, Xi S, Xu W, Young A H, Zandi P, Zhang P, Zöllner S, eQTLGen Consortium, BIOS Consortium, Adolfsson R, Agartz I, Alda M, Backlund L, Baune B T, Bellivier F, Berrettini W H, Biernacka J M, Blackwood D H R, Boehnke M, Børglum A D, Corvin A, Craddock N, Daly M J, Dannlowski U, Esko T, Etain B, Frye M, Fullerton J M, Gershon E S, Gill M, Goes F, Grigoroiu-Serbanescu M, Hauser J, Hougaard D M, Hultman C M, Jones I, Jones L A, Kahn R S, Kirov G, Landén M, Leboyer M, Lewis C M, Li Q S, Lissowska J, Martin N G, Mayoral F, McElroy S L, McIntosh A M, McMahon F J, Melle I, Metspalu A, Mitchell P B, Morken G, Mors O, Mortensen P B, Müller-Myhsok B, Myers R M, Neale B M, Nimgaonkar V, Nordentoft M, Nöthen M M, O'Donovan M C, Oedegaard K J, Owen M J, Paciga S A, Pato C, Pato M T, Posthuma D, Ramos-Quiroga J A, Ribasés M, Rietschel M, Rouleau G A, Schalling M, Schofield P R, Schulze T G, Serretti A, Smoller J W, Stefansson H, Stefansson K, Stordal E, Sullivan P F, Turecki G, Vaaler A E, Vieta E, Vincent J B, Werge T, Nurnberger J I, Wray N R, Di Florio A, Edenberg H J, Cichon S, Ophoff R A, Scott L J, Andreassen O A, Kelsoe J, Sklar P, Bipolar Disorder Working Group of the Psychiatric Genomics Consortium. 2019. Genome-wide association study identifies 30 loci associated with bipolar disorder. Nature Genetics, 51(5): 793-803 [DOI:10.1038/s41588-019-0397-8]
-
Sui J, Qi S L, van Erp T G M, Bustillo J, Jiang R T, Lin D D, Turner J A, Damaraju E, Mayer A R, Cui Y, Fu Z N, Du Y H, Chen J Y, Potkin S G, Preda A, Mathalon D H, Ford J M, Voyvodic J, Mueller B A, Belger A, McEwen S C, O'Leary D S, McMahon A, Jiang T Z, Calhoun V D. 2018. Multimodal neuromarkers in schizophrenia via cognition-guided MRI fusion. Nature Communications, 9(1): #3028 [DOI:10.1038/s41467-018-05432-w]
-
Tan J, Yuan S X, Ming W L, Sun X. 2018. Progress on analytical methods of imaging genomics. Current Biotechnology, 8(4): 277-283, 369 (谭俊, 袁少勋, 明文龙, 孙啸. 2018. 影像基因组学分析方法研究进展. 生物技术进展, 8(4): 277-283, 369) [DOI:10.19586/j.2095-2341.2018.0045]
-
Tian Y, Margulies D S, Breakspear M, Zalesky A. 2020. Topographic organization of the human subcortex unveiled with functional connectivity gradients. Nature Neuroscience, 23(11): 1421-1432 [DOI:10.1038/s41593-020-00711-6]
-
van den Heuvel M P, Scholtens L H, Kahn R S. 2019. Multiscale neuroscience of psychiatric disorders. Biological Psychiatry, 86(7): 512-522 [DOI:10.1016/j.biopsych.2019.05.015]
-
Van Essen D C, Smith S M, Barch D M, Behrens T E J, Yacoub E, Ugurbil K, WU-Minn HCP Consortium. 2013. The WU-Minn human connectome project: an overview. NeuroImage, 80: 62-79 [DOI:10.1016/j.neuroimage.2013.05.041]
-
Vázquez-Rodríguez B, Suárez L E, Markello R D, Shafiei G, Paquola C, Hagmann P, van den Heuvel M P, Bernhardt B C, Spreng R N, Misic B. 2019. Gradients of structure-function tethering across neocortex. Proceedings of the National Academy of Sciences of the United States of America, 116(42): 21219-21227 [DOI:10.1073/pnas.1903403116]
-
Wang D X, Eraslan B, Wieland T, Hallström B, Hopf T, Zolg D P, Zecha J, Asplund A, Li L H, Meng C, Frejno M, Schmidt T, Schnatbaum K, Wilhelm M, Ponten F, Uhlen M, Gagneur J, Hahne H, Kuster B. 2019. A deep proteome and transcriptome abundance atlas of 29 healthy human tissues. Molecular Systems Biology, 15(2): #8503 [DOI:10.15252/msb.20188503]
-
Wang H Y, Fan L Z, Song M, Liu B, Wu D Y, Jiang R T, Li J, Li A, Banaschewski T, Bokde A L W, Quinlan E B, Desrivières S, Flor H, Grigis A, Garavan H, Chaarani B, Gowland P, Heinz A, Ittermann B, Martinot J L, Martinot M L P, Artiges E, Nees F, Orfanos D P, Poustka L, Millenet S, Fröhner J H, Smolka M N, Walter H, Whelan R, Schumann G, Jiang T Z. 2021. Functional connectivity predicts individual development of inhibitory control during adolescence. Cerebral Cortex, 31(5): 2686-2700 [DOI:10.1093/cercor/bhaa383]
-
Wang Q X, Ding S L, Li Y, Royall J, Feng D, Lesnar P, Graddis N, Naeemi M, Facer B, Ho A, Dolbeare T, Blanchard B, Dee N, Wakeman W, Hirokawa K E, Szafer A, Sunkin S M, Oh S W, Bernard A, Phillips J W, Hawrylycz M, Koch C, Zeng H K, Harris J A, Ng L. 2020. The Allen mouse brain common coordinate framework: a 3D reference atlas. Cell, 181(4): 936-953 [DOI:10.1016/j.cell.2020.04.007]
-
Wingo T S, Liu Y, Gerasimov E S, Gockley J, Logsdon B A, Duong D M, Dammer E B, Lori A, Kim P J, Ressler K J, Beach T G, Reiman E M, Epstein M P, De Jager P L, Lah J J, Bennett D A, Seyfried N T, Levey A I, Wingo A P. 2021. Brain proteome-wide association study implicates novel proteins in depression pathogenesis. Nature Neuroscience, 24(6): 810-817 [DOI:10.1038/s41593-021-00832-6]
-
Wray N R, Ripke S, Mattheisen M, Trzaskowski M, Byrne E M, Abdellaoui A, Adams M J, Agerbo E, Air T M, Andlauer T M F, Bacanu S A, Bækvad-Hansen M, Beekman A F T, Bigdeli T B, Binder E B, Blackwood D R H, Bryois J, Buttenschøn H N, Bybjerg-Grauholm J, Cai N, Castelao E, Christensen J H, Clarke T K, Coleman J I R, Colodro-Conde L, Couvy-Duchesne B, Craddock N, Crawford G E, Crowley C A, Dashti H S, Davies G, Deary I J, Degenhardt F, Derks E M, Direk N, Dolan C V, Dunn E C, Eley T C, Eriksson N, Escott-Price V, Kiadeh F H F, Finucane H K, Forstner A J, Frank J, Gaspar H A, Gill M, Giusti-Rodríguez P, Goes F S, Gordon S D, Grove J, Hall L S, Hannon E, Hansen C S, Hansen T F, Herms S, Hickie I B, Hoffmann P, Homuth G, Horn C, Hottenga J J, Hougaard D M, Hu M, Hyde C L, Ising M, Jansen R, Jin F L, Jorgenson E, Knowles J A, Kohane I S, Kraft J, Kretzschmar W W, Krogh J, Kutalik Z, Lane J M, Li Y H, Li Y, Lind P A, Liu X X, Lu L N, MacIntyre D J, MacKinnon D F, Maier R M, Maier W, Marchini J, Mbarek H, McGrath P, McGuffin P, Medland S E, Mehta D, Middeldorp C M, Mihailov E, Milaneschi Y, Milani L, Mill J, Mondimore F M, Montgomery G W, Mostafavi S, Mullins N, Nauck M, Ng B, Nivard M G, Nyholt D R, O'Reilly P F, Oskarsson H, Owen M J, Painter J N, Pedersen C B, Pedersen M G, Peterson R E, Pettersson E, Peyrot W J, Pistis G, Posthuma D, Purcell S M, Quiroz J A, Qvist P, Rice J P, Riley B P, Rivera M, Saeed Mirza S, Saxena R, Schoevers R, Schulte E C, Shen L, Shi J X, Shyn S I, Sigurdsson E, Sinnamon G B C, Smit J H, Smith D J, Stefansson H, Steinberg S, Stockmeier C A, Streit F, Strohmaier J, Tansey K E, Teismann H, Teumer A, Thompson W, Thomson P A, Thorgeirsson T E, Tian C, Traylor M, Treutlein J, Trubetskoy V, Uitterlinden A G, Umbricht D, Van der Auwera S, van Hemert A M, Viktorin A, Visscher P M, Wang Y, Webb B T, Weinsheimer S M, Wellmann J, Willemsen G, Witt S H, Wu Y, Xi H S, Yang J, Zhang F T, eQTLGen, 23andMe, Arolt V, Baune B T, Berger K, Boomsma D I, Cichon S, Dannlowski U, de Geus E C J, DePaulo J R, Domenici E, Domschke K, Esko T, Grabe H J, Hamilton S P, Hayward C, Heath A C, Hinds D A, Kendler K S, Kloiber S, Lewis G, Li Q S, Lucae S, Madden P F A, Magnusson P K, Martin N G, McIntosh A M, Metspalu A, Mors O, Mortensen P B, Müller-Myhsok B, Nordentoft M, Nöthen M M, O'Donovan M C, Paciga S A, Pedersen N L, Penninx B, Perlis R H, Porteous D J, Potash J B, Preisig M, Rietschel M, Schaefer C, Schulze T G, Smoller J W, Stefansson K, Tiemeier H, Uher R, Völzke H, Weissman M M, Werge T, Winslow A R, Lewis C M, Levinson D F, Breen G, Børglum A D, Sullivan P F, Major Depressive Disorder Working Group of the Psychiatric Genomics Consortium. 2018. Genome-wide association analyses identify 44 risk variants and refine the genetic architecture of major depression. Nature Genetics, 50(5): 668-681 [DOI:10.1038/s41588-018-0090-3]
-
Wu D Y, Fan L Z, Song M, Wang H Y, Chu C Y, Yu S, Jiang T Z. 2020. Hierarchy of connectivity-function relationship of the human cortex revealed through predicting activity across functional domains. Cerebral Cortex, 30(8): 4607-4616 [DOI:10.1093/cercor/bhaa063]
-
Xia M R, He Y. 2018. Psychoradiology big data and human connectomics. Chinese Journal of Psychiatry, 51(4): 221-223 (夏明睿, 贺永. 2018. 精神影像大数据与人脑连接组学. 中华精神科杂志, 51(4): 221-223) [DOI:10.3760/cma.j.issn.1006-7884.2018.04.001]
-
Xu F, Shen Y, Ding L F, Yang C Y, Tan H, Wang H, Zhu Q Y, Xu R, Wu F Y, Xiao Y Y, Xu C, Li Q W, Su P, Zhang L I, Dong H W, Desimone R, Xu F Q, Hu X T, Lau P M, Bi G Q. 2021. High-throughput mapping of a whole rhesus monkey brain at micrometer resolution. Nature Biotechnology, 39(12): 1521-1528 [DOI:10.1038/s41587-021-00986-5]
-
Xu Q, Guo L N, Cheng J L, Wang M Y, Geng Z J, Zhu W Z, Zhang B, Liao W H, Qiu S J, Zhang H, Xu X J, Yu Y Q, Gao B, Han T, Yao Z W, Cui G B, Liu F, Qin W, Zhang Q, Li M J, Liang M, Chen F, Xian J F, Li J C, Zhang J, Zuo X N, Wang D W, Shen W, Miao Y W, Yuan F, Lui S, Zhang X C, Xu K, Zhang L J, Ye Z X, Yu C S, CHIMGEN Consortium. 2020. CHIMGEN: a Chinese imaging genetics cohort to enhance cross-ethnic and cross-geographic brain research. Molecular Psychiatry, 25(3): 517-529 [DOI:10.1038/s41380-019-0627-6]
-
Yao D R, Sui J, Wang M L, Yang E K, Jiaerken Y, Luo N, Yap P T, Liu M X, Shen D G. 2021a. A mutual multi-scale triplet graph convolutional network for classification of brain disorders using functional or structural connectivity. IEEE transactions on medical imaging, 40(4): 1279-1289 [DOI:10.1109/tmi.2021.3051604]
-
Yao Z Z, Liu H Q, Xie F M, Fischer S, Adkins R S, Aldridge A I, Ament S A, Bartlett A, Behrens M M, Van den Bérge K, Bertagnolli D, de Bezieux H R, Biancalani T, Booeshaghi A S, Bravo H C, Casper T, Colantuoni C, Crabtree J, Creasy H, Crichton K, Crow M, Dee N, Dougherty E L, Doyle W I, Dudoit S, Fang R X, Felix V, Fong O, Giglio M, Goldy J, Hawrylycz M, Herb B R, Hertzano R, Hou X M, Hu Q W, Kancherla J, Kroll M, Lathia K, Li Y E, Lucero J D, Luo C Y, Mahurkar A, McMillen D, Nadaf N M, Nery J R, Nguyen T N, Niu S Y, Ntranos V, Orvis J, Osteen J K, Pham T, Pinto-Duarte A, Poirion O, Preissl S, Purdom E, Rimorin C, Risso D, Rivkin A C, Smith K, Street K, Sulc J, Svensson V, Tieu M, Torkelson A, Tung H, Vaishnav E D, Vanderburg C R, van Velthoven C, Wang X X, White O R, Huang Z J, Kharchenko P V, Pachter L, Ngai J, Regev A, Tasic B, Welch J D, Gillis J, Macosko E Z, Ren B, Ecker J R, Zeng H K, Mukamel E A. 2021b. A transcriptomic and epigenomic cell atlas of the mouse primary motor cortex. Nature, 598(7879): 103-110 [DOI:10.1038/s41586-021-03500-8]
-
Yi C L, Yao R W, Song L Y, Jiang L, Si Y J, Li P Y, Li F L, Yao D Z, Zhang Y, Xu P. 2021. A novel method for constructing EEG large-scale cortical dynamical functional network connectivity (dFNC): WTCS. IEEE Transactions on Cybernetics: #3090770 [DOI:10.1109/TCYB.2021.3090770]
-
Zhang D Q, Shen D G, Alzheimer's Disease Neuroimaging Initiative. 2012. Multi-modal multi-task learning for joint prediction of multiple regression and classification variables in Alzheimer's disease. NeuroImage, 59(2): 895-907 [DOI:10.1016/j.neuroimage.2011.09.069]
-
Zhao B X, Li T F, Yang Y, Wang X F, Luo T Y, Shan Y, Zhu Z L, Xiong D, Hauberg M E, Bendl J, Fullard J F, Roussos P, Li Y, Stein J L, Zhu H T. 2021. Common genetic variation influencing human white matter microstructure. Science, 372(6548): #3736 [DOI:10.1126/science.abf3736]
-
Zhong S J, Ding W Y, Sun L, Lu Y F, Dong H, Fan X Y, Liu Z Y, Chen R G, Zhang S, Ma Q, Tang F C, Wu Q, Wang X Q. 2020. Decoding the development of the human hippocampus. Nature, 577(7791): 531-536 [DOI:10.1038/s41586-019-1917-5]
-
Zong W J, Wu R L, Chen S Y, Wu J J, Wang H B, Zhao Z, Chen G Q, Tu R, Wu D L, Hu Y H, Xu Y Y, Wang Y, Duan Z L, Wu H T, Zhang Y F, Zhang J, Wang A M, Chen L Y, Cheng H P. 2021. Miniature two-photon microscopy for enlarged field-of-view, multi-plane and long-term brain imaging. Nature Methods, 18(1): 46-49 [DOI:10.1038/s41592-020-01024-z]
-
Zuo N M, Jiang T Z. 2020. Multi-modal human brain mapping and Brainnetome study. Scientia Sinica Vitae, 50(11): 1296-1308 (左年明, 蒋田仔. 2020. 多模态人脑影像与脑网络组学研究. 中国科学(生命科学), 50(11): 1296-1308) [DOI:10.1360/SSV-2019-0228]